

【原创】清华大学王宏伟教授: 冷冻电镜的发展意味着结构生物学的终结?
日期:2017-02-20 浏览次数:9548
【编者按】近年来冷冻电镜技术的发展非常迅猛。软件与硬件发展的完美结合,使得利用冷冻电镜进行生物大分子结构解析的分辨率和效率都大大提高,有关方法学突破和具有里程碑意义的重要结构解析结果也层出不穷。最近Nature Methods 上发表的文章 “cryoSPARC: algorithms for rapid unsupervised cryo-EM structure determination”(点击这里看该文的解读),在年轻学子中引起了又一波关于从事冷冻电镜的研究者是否很快失业的讨论。
为此,中国生物物理学会特邀请冷冻电镜方面的专家、清华大学生命学院院长、北京结构生物学高精尖创新中心副主任王宏伟教授(将担任 3D EM- Gordon Research Conference 2018年副主席,2019年主席,详情请点击左下方 阅读原文)撰文,回顾冷冻电镜的发展历程和目前的发展态势,并分析了冷冻电镜技术中尚未解决的重要问题,以及未来发展的趋势。期待本文能够帮助结构生物学领域的年轻学人及时调整心态和知识结构,适应甚至引领冷冻电镜领域的发展和变化。

王宏伟教授(图片来自清华大学官网)
最近,我第50次收到邮件咨询应该如何建一个最新型的冷冻电镜设施。今天,如果你有机会问一个世界知名大学的校长:“您的学校生命学科发展有哪些最需要的设备?”冷冻电镜很可能是位居首位的大型设备之一,当然前提是学校有足够的经费来支付昂贵的设备采购、安装与运行费用。与此形成鲜明对比的是,5年前,相当多的大学还在考虑是否应该在本校设立冷冻电镜这个研究领域;10年前,绝大多数的学校可能还不知道居然有一种电子显微镜要用“冷冻”来做定语。
现代生命科学的发展速度飞快,某些领域的发展甚至可以用“日新月异”来形容。冷冻电镜技术在过去3年来的表现绝对配得上这个词。利用冷冻电镜进行生物大分子结构解析,不需要大量样品,不需要结晶。其解析结果的分辨率不断刷新世界纪录,结构解析的诸多算法层出不穷,结构解析效率也迅速提高。很多科学家苦苦钻研了十几年乃至几十年的复杂结构很快被解析出来,如TRPV channel,spliceosome,calcium channel,respirasome,dynactin等。现在,几乎所有的结构生物学家都在试图学习冷冻电镜技术,或者与冷冻电镜实验室开展密切的合作。
如今,全世界所有的冷冻电镜平台都人满为患。高端仪器设备的供不应求已经成为结构生物学家抱怨最多的问题之一了,甚至可能比对经费申请困难的抱怨还要多。5年前,我们建设清华冷冻电镜平台时曾有人担心:“这么多的高端冷冻电镜(3台300kV电镜),未来会有足够的人使用吗?它们的运行费用将如何维持?”今天,我们遗憾的是当初没有规划更多的设备与空间,导致现在的运行机时非常紧张。虽然我们平台的运行效率已远远高于设计规划,却仍然无法满足日益增长的用户需求。目前,美国、英国、欧洲、日本都纷纷在建设或筹划建设国家级冷冻电镜中心,希望通过集中的平台资源为全世界的结构生物学家提供高效的数据采集与分析服务。能否以同步辐射光源的运行模式来实现冷冻电镜平台的运行,是当下的热点讨论话题。
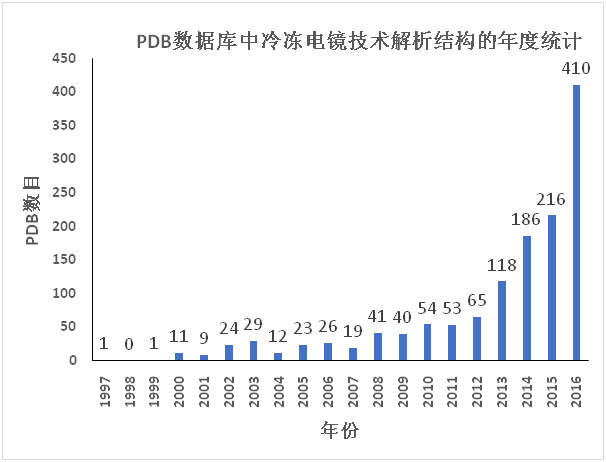
2016年是冷冻电镜技术解析出来的结构数目跳跃性增长的一年(数据来自www.rcsb.org)
冷冻电镜技术虽然在3年前一夜走红,却并非横空出世,在此之前,它已经发展了近半个世纪。上世纪60年代英国剑桥大学MRC分子生物学实验室的Aaron Klug领导的小组首次实现了从多个角度的二维电子显微图像获得三维结构,并因此获得了1982年的诺贝尔化学奖[1]。70年代美国加州大学伯克利分校的Robert Glaeser及其团队率先证明冷冻于液氮温度下的蛋白质分子可以在电子显微镜的高真空中保持在含水状态,并且可以耐受高能电子的辐照而仍然保持其精细结构[2]。与此同时,MRC分子生物学实验室的Richard Henderson和Nigel Unwin应用葡萄糖包埋的技术保持蛋白质二维晶体在真空中的含水状态,并首次用电子显微镜解析了细菌紫红质蛋白的7埃分辨率结构[3]。到80年代初,欧洲分子生物学实验室的Jacques Dubochet课题组实现了将溶液状态的生物大分子速冻在玻璃态的冰中,在液氮温度下的电子显微镜观察,从而奠定了冷冻电镜制样与观察的基本技术手段[4]。这也标志着冷冻电镜(Cryo-Electron Microscopy或 Electron Cryo-Microscopy)技术的诞生。
冷冻电镜技术多年的发展已经产生了多种解析生物分子或细胞结构的方法,现在备受瞩目的冷冻电镜结构解析则主要是指单颗粒三维重构技术。该方法通过对大量离散分布的单个分子的电子显微像进行统计分析来解析结构。这种方法最早由美国纽约州Wadsworth中心的Joachim Frank领导的课题组开发,从70年代开始,到90年代形成了比较完整的结构解析算法原理[5]。但是直到上个世纪末,冷冻电镜结构解析的主流方法还不是单颗粒三维重构技术,而是更为成熟的针对二维晶体和螺旋对称性结构的电子晶体学解析手段。这主要是因为单颗粒技术对电子显微镜图像的数量和计算机资源的需求都很大,当时的显微镜及计算机水平尚无法满足要求。进入21世纪,电子显微镜硬件的稳定性及自动化程度稳步提升,超大规模计算的能力也大幅度提高,为单颗粒三维重构技术解析生物大分子结构的有效性奠定了良好的基础,使开发新的结构解析算法成为可能。从2000年左右开始,多种单颗粒结构解析软件(如SPIDER,IMAGIC,EMAN,XMIPP,Frealign 等)各以其特有的优势在领域内得到应用,并大大地推动了单颗粒三维重构技术的发展。到2008-2009年,具有正二十面体对称性的病毒颗粒已经可以应用单颗粒冷冻电镜技术解析到原子分辨率,标志着这一技术进入了原子分辨率时代[6][7]。冷冻电镜领域的研究团队也日渐壮大起来。在上世纪80年代,从事冷冻电镜相关的科研课题组全世界不超过20个,总人数在100人左右;到本世纪初,冷冻电镜的研究团队有近50个;到2010年左右,以冷冻电镜为主的研究团队已经超过100个。冷冻电镜吸引了越来越多数学、物理学、计算机科学、工程学背景的科学家的加入,新的概念与新的技术被引入到这个领域中来。此时,革新性的技术突破已如箭在弦上。
2013年底,美国加州大学旧金山分校的程亦凡与David Julies组在Nature上合作发表了两篇论文,报道了TRPV1膜蛋白的原子分辨率冷冻电镜结构,在整个结构生物学领域一石激起千层浪[8][9]。几乎在一夜之间,X射线晶体学界的著名科学家都将他们的目光投向了冷冻电镜技术。在铺天盖地的报道中,很多生物学家第一次听说冷冻电镜,觉得这带来了结构生物学的革命。在冷冻电镜领域的人看来,这也确实是一次革命,是单颗粒结构解析分辨率的革命,而且早在2011-2012年间就已经开始了。一种新的直接电子探测成像设备的发明及应用在2011年问世,当即在冷冻电镜领域内引起了不小的轰动[10]。2013年初,程亦凡课题组和英国MRC分子生物学实验室的SjorsScheres课题组独立地发表论文,证明应用直接电子探测成像设备并结合新的图像处理工具可以解析生物大分子的原子分辨率结构[11][12]。这种新型的成像设备大大提高了冷冻电镜照片的质量,使原来无法被观察到的结构细节变得清晰。与此同时,更为有效的图像处理与结构解析算法被开发出来。SjorsScheres开发的基于概率统计分析的算法Relion能更有效地处理低信噪比的图像,成为单颗粒结构解析的利器[13][14]。当然,这也得益于高性能计算的迅速发展。
软件与硬件发展的完美结合,促成了一次技术上的飞跃。整个冷冻电镜领域对于这次革命的到来都有些准备不足。所有安装了新硬件、使用新算法的实验室都发现图像存储资源、网络传输速度、计算资源成为新技术使用的主要瓶颈。较早用上了新硬件、新软件的幸运儿,在解析出了高分辨率的结构后,发现模型的搭建与分析是自己并不擅长的技术。与此同时,以X射线晶体学为主要研究手段的结构生物学实验室开始应用冷冻电镜技术,很快以他们在生物化学和高分辨率结构模型搭建中的丰富经验后来居上,超过此前一直以冷冻电镜为主要研究手段的课题组。更重要的,新的技术意味着旧有的经验与观念的失效——无论对冷冻电镜图像的评估,还是对结构解析过程的分析,都需要有新的评判标准。而整个冷冻电镜领域则必须对技术革命所带来的新的可能发展方向做更广泛的探索和深入的研讨。过去几年来的多个冷冻电镜国际会议非常激动人心,也令人心惊胆战。几乎每隔几个月,就会有人报道新的方法学突破和具有里程碑意义的重要结构解析结果。
在短短的3年时间里,冷冻电镜已经成为生物大分子结构解析的主要方法。冷冻电镜的用户群体正在以几何级数的规模增长。很多不同专业背景的研究人员纷纷加入冷冻电镜技术方法开发的行列中。冷冻电镜单颗粒三维重构的技术正在变得越来越成熟。包括相位板、能量过滤器、物镜球差矫正装置等一系列新硬件设备的应用开发进一步推进冷冻电镜技术的极限;新的软件算法不断涌现,处理效率越来越高,用户界面越来越友好;冷冻电镜数据采集的自动化程度越来越高,操作冷冻电镜设备的技术门槛越来越低。最新的成果包括:对HIV病毒衣壳前体可以通过Tomography与单颗粒技术的结合解析到3.9埃的近原子分辨率[15];应用相位板冷冻电镜单颗粒技术可以解析60kDa分子量的血红蛋白高分辨率结构[16];一种基于随机梯度下降算法的cryoSPARC程序已经可以在单机工作站上运行,初步测试结果表明效率比Relion高分辨率结构解析有数量级的提升,而且不需要初始模型[17]。在冷冻电镜单颗粒技术中,曾经是结构生物学主要限速步骤的晶体生长和筛选过程完全免除,因此大大提高了结构解析的效率。在比较快的例子里,从获得完全未知结构的蛋白分子到解析出结构只需要不到1个月的时间。以至于有人感慨:冷冻电镜使结构生物学越来越大众化了,从事冷冻电镜研究的人将很快失去工作,甚至结构生物学也将面临终结。这种论断却未免把冷冻电镜的潜力看得太浅了,也误解了结构生物学的真谛。
与X射线晶体学相比,电子显微镜作为结构生物学的手段至少晚诞生了20年,冷冻电镜作为较常规的高分辨率结构生物学的解析手段则晚了近30年。冷冻电镜今天的发展还只是刚刚开始,还有很多重要技术问题尚未解决。比如:
1、在单颗粒分析技术中,如何将生物大分子机器的结构变化及不同构象的分布情况进行精确的描述?如何获得生化反应过程中所有步骤的生物大分子结构信息?这将会提供静态结构以外的重要信息,如分子机器的热力学或动力学的相关信息。
2、如何进一步提高冷冻电镜结构解析的分辨率,达到1埃以上的超高分辨率?这将有利于对分子中的电势分布做更精细的解析,从而更好地理解生物大分子机器的化学本质乃至量子本质。
3、如何获得更接近生理状态的生物大分子结构,如何在细胞乃至组织的原位获得高精度的结构信息?这将实现结构生物学与细胞生物学的融合,甚至可能发展出新的医学检测手段。
4、如何将冷冻电镜技术与其它技术手段如质谱技术、测序技术、超高分辨率光学成像技术、单分子操控技术等整合起来,开拓新的方法学领域?在广义的生命科学研究中,未来的电子显微镜技术方法还可能用来分析生物样品,获得更多复杂的生物学信息,届时结构生物学家要面对的挑战是如何更有效地整合、挖掘、展示海量的结构信息,全方位地了解我们面对的研究对象。
未来几年里,冷冻电镜单颗粒技术的发展将日臻成熟,建立国家级的冷冻电镜中心,集中高效地提供高质量的冷冻电镜数据收集与分析的服务,将成为可能,也是必然的发展趋势。但以科研课题组为依托,开展新技术开发与应用的研究,将继续作为冷冻电镜领域的主流。新的硬件设备、软件算法会不断被开发出来,帮助生物学家看到更多以前无法想象的结构信息。这将始终是冷冻电镜领域的主旋律。随着技术的进步,结构生物学也将遵循半个多世纪以来的发展轨迹,在结构解析的质量与效率、在研究对象的复杂程度、在探讨生物学问题的深度和广度等方面,提出更高的要求。结构作为功能的基础和生命现象的载体,揭示生命体中原子、分子、细胞、组织、器官等各层级的空间组织方式、变化规律及其与功能的关系,这些始终是结构生物学所关注的根本问题。新的技术进步势必为我们解析多种层级的结构信息提供更为有效的工具,把我们从繁杂的技术细节中解放出来,从而深入地思考生命现象的本质。结构生物学家应该及时调整自己的心态、知识结构、问题导向、研究方法,来适应甚至引领新的变化,正如在过去3年里很多结构生物学大师所做的那样。
致谢:感谢程亦凡、范潇、刘文楠、颜宁、张佩君对本文撰写提供的建议与补充。
参考文献:
1. De Rosier D J, Klug A. Reconstruction of threedimensional structures from electron micrographs. Nature, 1968, 217: 130-134.
2. Taylor K A, Glaeser R M. Electron diffraction of frozen, hydrated protein crystals.Science, 1974, 186: 1036–1037.
3. HendersonR, Unwin P N. Three-dimensional model of purple membrane obtained by electron microscopy. Nature, 1975, 257: 28-32.
4. DubochetJ, Lepault J, Freeman R, et al. Electron microscopy of frozen water and aqueous solutions. Journal of Microscopy, 1982, 128(3): 219-237.
5. Frank,J. Three-Dimensional Electron Microscopy of Macromolecular Assemblies. AcademicPress, San Diego, 1996.
6. Zhang X, Settembre E, Xu C, et al. Near-atomic resolution using electron cryomicroscopy and single-particle reconstruction. Proceedings of the National Academy of Sciences, 2008, 105(6): 1867-1872.
7. YuX, Jin L, Zhou Z H. 3.88 Å structure of cytoplasmic polyhedrosis virus bycryo-electron microscopy. Nature, 2008, 453(7193): 415-419.
8. LiaoM, Cao E, Julius D, et al. Structure of the TRPV1 ion channel determined by electron cryo-microscopy. Nature, 2013, 504(7478): 107-112.
9. CaoE, Liao M, Cheng Y, et al. TRPV1 structures in distinct conformations revealactivation mechanisms. Nature, 2013, 504(7478): 113-118.
10. Milazzo A C, Cheng A, Moeller A, et al. Initial evaluation of a direct detection device detector for single particle cryo-electron microscopy. Journal of structural biology, 2011, 176(3): 404-408.
11. LiX, Mooney P, Zheng S, et al. Electron counting and beam-induced motion correction enable near-atomic-resolution single-particle cryo-EM. Nature methods, 2013, 10(6): 584-590.
12. Bai X, Fernandez I S, McMullan G, et al. Ribosome structures to near-atomicresolution from thirty thousand cryo-EM particles. Elife, 2013, 2: e00461.
13. ScheresS H W. A Bayesian view on cryo-EM structure determination. Journal of molecular biology, 2012, 415(2): 406-418.
14. Scheres S H W. RELION: implementation of a Bayesian approach to cryo-EM structure determination. Journal of structural biology, 2012, 180(3): 519-530.
15. Schur F K M, Obr M, Hagen W J H, et al. An atomic model of HIV-1 capsid-SP1 reveals structures regulating assembly and maturation. Science, 2016, 353(6298): 506-508.
16. Khoshouei M, Radjainia M, Baumeister W, et al. Cryo-EM structure of haemoglobin at 3.2 Adetermined with the Volta phase plate. bioRxiv, 2016: 087841.
17. Punjani A, Rubinstein J L, Fleet D J, et al. cryoSPARC: algorithms for rapid unsupervised cryo-EM structure determination. Nature Methods, 2017.