张宏团队揭示SARS-CoV-2抑制自噬溶酶体形成的机制
日期:2020-12-22 浏览次数:15329
来源:BioArt
细胞自噬(autophagy)是一种在真核生物中高度保守的由溶酶体介导的胞内降解途径。自噬通过形成双层膜的自噬体包裹胞质中的待降解组分,随即运输至溶酶体进行降解。细胞自噬对于细胞应对各种应激条件以及维持稳态平衡至关重要,并且能够参与抵抗外来入侵的病原菌和病毒。在病毒侵染的过程中,一些病毒蛋白以及一系列病毒感染引起的细胞应激均可激活宿主细胞的自噬通路【1-3】。自噬作为细胞的防御机制,将病毒或病毒蛋白运送到溶酶体进行降解,激活先天性和适应性免疫反应,或调节病毒诱导的细胞死亡,从而抵御病毒感染【4,5】。
病毒也进化出各种机制以逃避自噬降解,一些病毒甚至能够利用自噬通路的膜结构完成自身的复制和释放【6】。比如多种正链RNA病毒能够阻断自噬体与溶酶体的融合,并将RNA复制机器锚定在自噬膜结构上进行病毒基因组的复制【7-9】。而在MHV,MERS-CoV和SARS-CoV等冠状病毒的侵染过程中,其复制和转录复合物(RTCs)能够锚定在细胞内的双层膜囊泡(DMV)上行使功能,而复制形成的病毒RNA产物被储存于DMV中【10,11】。这些病毒RNA能够通过跨双膜的孔道被运输到细胞质中进行翻译或病毒组装【12】。研究表明冠状病毒侵染细胞中,DMV的形成并不需要自噬机器的参与。而最近的一项研究表明,β家族的冠状病毒(包括新冠病毒等)通过晚期内吞体/溶酶体从细胞中释放新组装的病毒颗粒【13】。目前对SARS-CoV-2如何改变细胞的内膜系统以及自噬活性的机制仍知之甚少。因此阐明SARS-CoV-2编码的蛋白质与宿主因子的相互作用机制,特别是对于在其他冠状病毒中不保守的辅助蛋白的研究,对于我们理解SARS-CoV-2的高传染性和致病性以及研发潜在的COVID-19治疗方法至关重要。
2020年12月16日,中国科学院生物物理研究所张宏课题组在Developmental Cell杂志在线发表了题为“ORF3a of the COVID-19 virus SARS-CoV-2 blocks HOPS complex-mediated assembly of the SNARE complex required for autolysosome formation”的研究论文,该文揭示了SARS-CoV-2编码的辅助蛋白ORF3a通过阻断HOPS复合物介导的SNARE复合体的组装,从而抑制自噬溶酶体的形成。ORF3a还能够抑制溶酶体的功能,并导致溶酶体损伤。此外, SARS-CoV-2病毒感染也能够导致VPS39亚基在晚期内吞体上聚集,从而阻断自噬溶酶体的形成,抑制自噬活性。
研究发现,在细胞中表达ORF3a后会导致自噬底物p62和自噬标志分子LC3大量累积。但与对照细胞相比,表达ORF3a的细胞中,LC3与LAMP1(晚期内吞体/溶酶体的标记物)的共定位会显著降低。此外,电镜结果也表明ORF3a表达细胞中,有降解活性的自噬溶酶体的形成受到抑制。
进一步研究发现ORF3a会影响HOPS复合物的亚细胞定位。ORF3a主要定位于晚期内吞体/溶酶体上(图1A),且与HOPS复合物的亚基VPS39直接互作。在对照组细胞中,VPS39主要定位在细胞质中(图1B);而在表达ORF3a的细胞中,VPS39-GFP形成大量的点状结构,并与ORF3a和RAB7共定位(图1C)。ORF3a会干扰功能性的HOPS复合物的形成:表达ORF3a会增强VPS39 与其它HOPS亚基VPS18或VPS11的相互作用,但减弱VPS39与亚复合物VPS3A/VPS16的相互作用。ORF3a干扰HOPS复合物与SNARE蛋白STX17的相互作用,并抑制SNARE复合物(STX17-SNAP29-VAMP8)的组装。
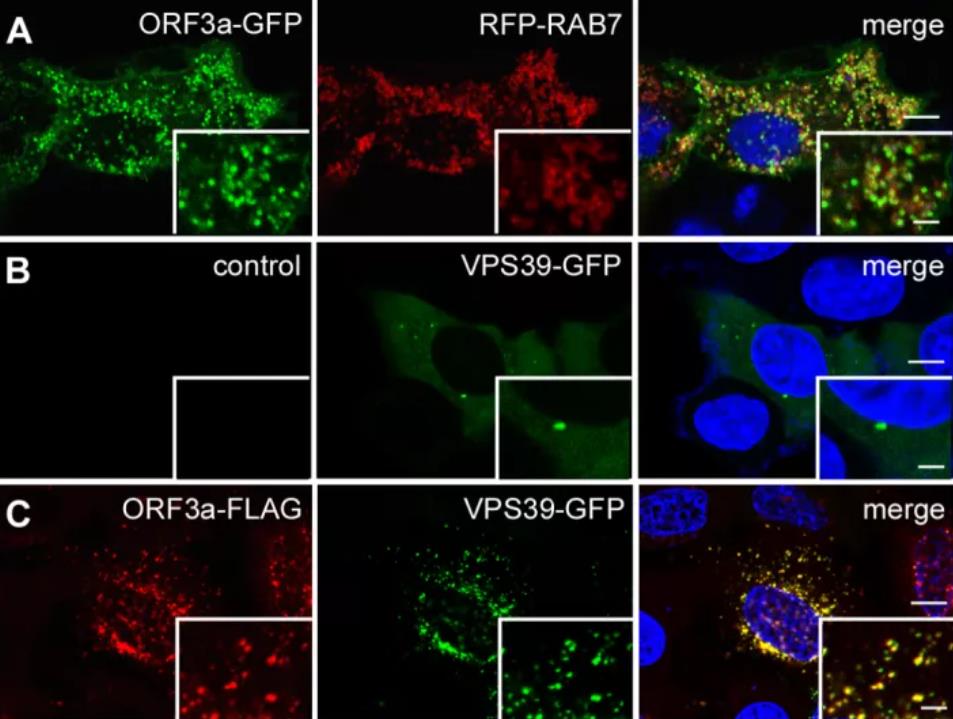
图1. ORF3a 定位于晚期内吞体,并导致VPS39 在晚期内吞体聚集
该研究还发现ORF3a可以抑制溶酶体功能并引起溶酶体损伤。ORF3a表达的细胞中,用于检测溶酶体降解活性的DQ-BSA所形成的荧光斑点的数量远低于对照细胞(图2A-2C)。此外,表达ORF3a的细胞中,特异性标记受损的内吞体/溶酶体的标记物Galectin 3形成更多的斑点结构(图2D-2F)。这些结果表明,表达ORF3a会干扰溶酶体功能,并引起溶酶体损伤。
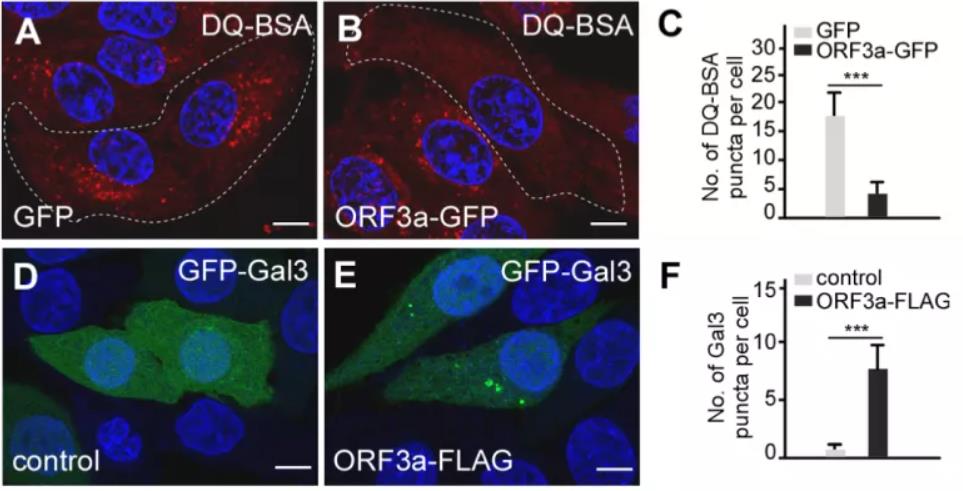
图2. ORF3a 表达干扰溶酶体功能,引起溶酶体损伤
最后,研究者进行了SARS-CoV-2病毒感染实验,发现病毒感染可阻断细胞自噬活性(图3A),导致自噬体结构的累积。与表达ORF3a蛋白相似,SARS-CoV-2病毒感染会导致HOPS亚基VPS39在晚期内吞体上聚集(图3B, 3C),并干扰HOPS复合物的形成。
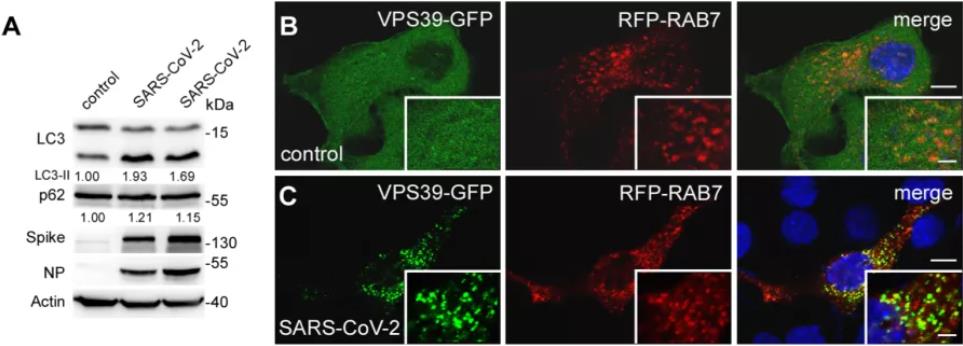
图3. SARS-CoV-2病毒感染抑制自噬活性,导致VPS39在晚期内吞体聚集
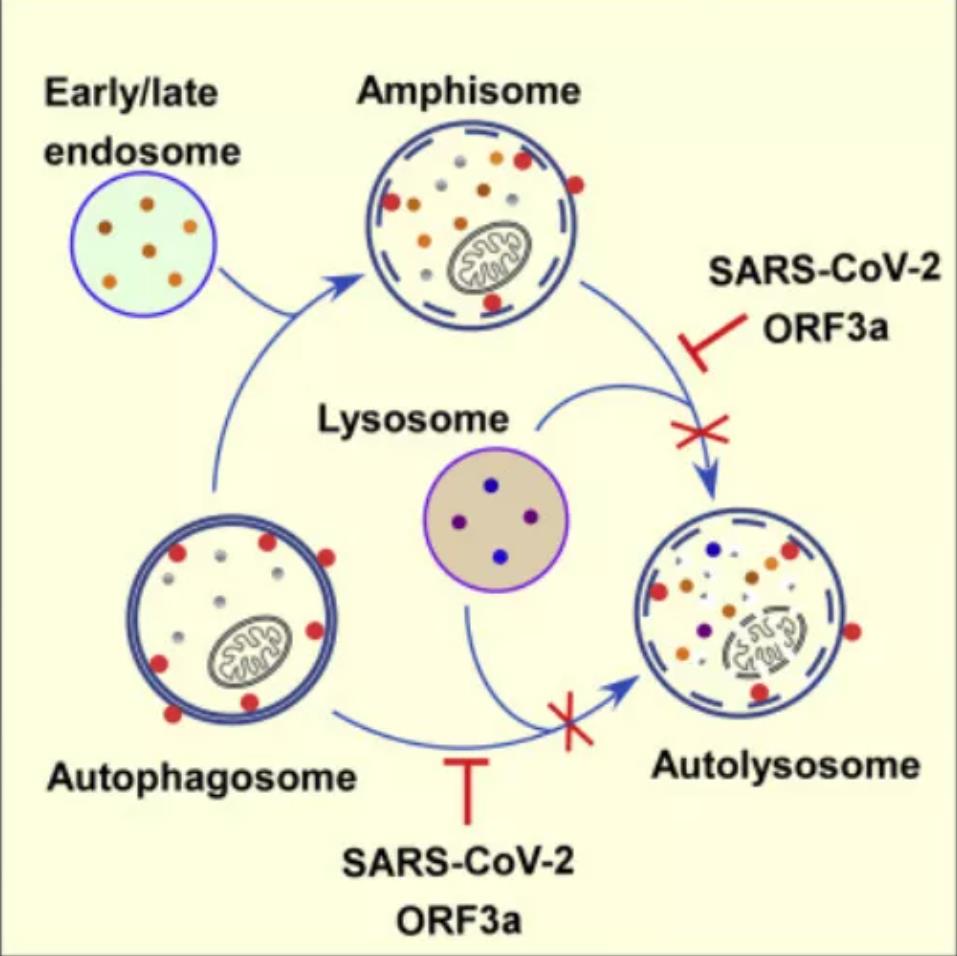
综上,该研究揭示了COVID-19病毒SARS-CoV-2的辅助蛋白ORF3a通过阻断自噬体与溶酶体的融合来抑制自噬活性,从而逃脱自噬的监控机制。这些研究对于我们理解SARS-CoV-2的高传染性和致病性以及开发潜在的COVID-19治疗方法有重要意义。
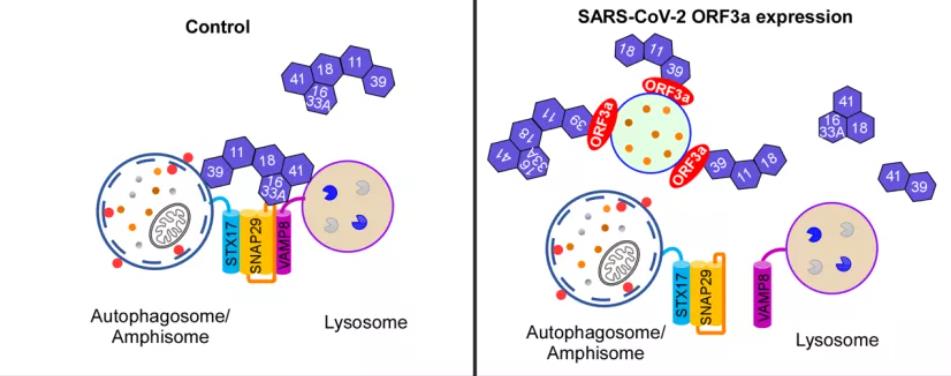
图4. ORF3a通过阻断HOPS复合物介导的SNARE复合体组装抑制自噬溶酶体的形成
原文链接:
https://doi.org/10.1016/j.devcel.2020.12.010
参考文献:
1. Dreux, M., and Chisari, F.V. (2010). Viruses and the autophagy machinery. Cell Cycle 9, 1295-1307.2. Jackson, W.T. (2015). Viruses and the autophagy pathway. Virology 479-480, 450-456.3. Viret, C., Rozieres, A., and Faure, M. (2018). Autophagy during Early Virus-Host Cell Interactions. J. Mol. Biol. 430, 1696-1713.4. Deretic, V., Saitoh, T., and Akira, S. (2013). Autophagy in infection, inflammation and immunity. Nat. Rev. Immunol. 13, 722-737.5. Levine, B., Mizushima, N., and Virgin, H.W. (2011). Autophagy in immunity and inflammation. Nature 469, 323-335.6. Choi, Y., Bowman, J.W., and Jung, J.U. (2018). Autophagy during viral infection - a double-edged sword. Nat. Rev. Microbiol. 16, 340-353.7. Ding, B., Zhang, G., Yang, X., Zhang, S., Chen, L., Yan, Q., Xu, M., Banerjee, A.K., and Chen, M. (2014). Phosphoprotein of human parainfluenza virus type 3 blocks autophagosome-lysosome fusion to increase virus production. Cell Host Microbe 15, 564-577.8. Corona, A.K., Saulsbery, H.M., Velazquez, A.F.C., and Jackson, W.T. (2018). Enteroviruses Remodel Autophagic Trafficking through Regulation of Host SNARE Proteins to Promote Virus Replication and Cell Exit. Cell Rep. 22, 3304-3314.9. Mohamud, Y., Shi, J.Y., Qu, J.Y., Poon, T., Xue, Y.C., Deng, H.Y., Zhang, J.C., and Luo, H.L. (2018). Enteroviral Infection Inhibits Autophagic Flux via Disruption of the SNARE Complex to Enhance Viral Replication. Cell Rep. 22, 3292-3303.10. Knoops, K., Kikkert, M., van den Worm, S.H.E., Zevenhoven-Dobbe, J.C., van der Meer, Y., Koster, A.J., Mommaas, A.M., and Snijder, E.J. (2008). SARS-coronavirus replication is supported by a reticulovesicular network of modified endoplasmic reticulum. Plos Biol. 6, 1957-1974.11. Snijder, E.J., Limpens, R.W.A.L., de Wilde, A.H., de Jong, A.W.M., Zevenhoven-Dobbe, J.C., Maier, H.J., Faas, F.F.G.A., Koster, A.J., and Barcena, M. (2020). A unifying structural and functional model of the coronavirus replication organelle: Tracking down RNA synthesis. Plos Biol. 18. e3000715.12. Wolff, G., Limpens, R.W.A.L., Zevenhoven-Dobbe, J.C., Laugks, U., Zheng, S., de Jong, A.W.M., Koning R.I., Agard, D.A., Grünewald, K., Koster, A.J., et al. (2020). A molecular pore spans the double membrane of the coronavirus replication organelle. Science 369, 1395-1398.13. Ghosh, S., Dellibovi-Ragheb, T. A., Kerviel, A., Pak, E., Qiu, Qi., Fisher, M., Takvorian, P.M., Bleck, C., Hsu, V., Fehr, A.R., et al. (2020). β-Coronaviruses Use Lysosomes for Egress Instead of the Biosynthetic Secretory Pathway. Cell S0092-8674, 31446-X.






