李国红课题组及合作者揭示了静息态基因的染色质结构特征并提出预测基因转录潜能的新方法
日期:2020-07-29 浏览次数:13243
作者:李国红研究组
来源:大屯路15号
2020年7月28日,国际学术期刊《Cell Reports》杂志在线发表了李国红课题组与暨南大学附属深圳市宝安区妇幼保健院许雪青课题组合作完成的研究论文"Analysis of Local Chromatin States Reveals Gene Transcription Potential during Mouse Neural Progenitor Cell Differentiation"。该研究工作解析了静息态基因的染色质结构特征,揭示了染色质状态与基因转录潜能之间的关系,并发现了一些参与神经元前体细胞发育的重要调控因子。
真核生物基因组DNA通过和组蛋白相互作用形成各种复杂的染色质结构而储存于细胞核中。作为遗传信息的载体,染色质是各种DNA相关生命活动等赖以进行并得以精准调控的底物,也是外界环境信号调控基因表达的窗口,其高级结构受到多种因素的动态调控。染色质结构的动态性和可塑性在基因转录沉默和激活过程中起重要作用,从而为表观遗传提供一个重要的信息整合平台。因此细胞基因组特定的染色质结构决定了细胞特定的基因转录谱和转录模式,从而决定细胞的组织特异性和细胞命运。根据染色质表观遗传标志以及染色质结合蛋白分布规律,染色质状态可以被分为许多不同的类型,如活跃状态、抑制状态以及静息态等。在众多的染色质状态中,静息态因为与干细胞的多能性相关,且在细胞分化过程中与基因快速激活的潜能密切关联,所以受到很多研究者的关注。在以往的研究中,静息态基因是通过同时存在两个相对立的染色质修饰来定义的,如表征激活状态的组蛋白H3第4位赖氨酸的三甲基化(H3K4me3)以及代表抑制状态的组蛋白H3第27位赖氨酸的三甲基化(H3K27me3)等等。然而,迄今为止,静息态染色质的结构特征还未见报道,且这种状态与基因转录潜能的关系还不甚清楚。该研究工作利用不同染色质结构状态对微球菌核酸酶(MNase)敏感性不同的特点,设计了基于时间梯度的温和MNase酶切实验(TC-rMNase-seq),通过对测序数据的分析发现,低紧密度染色质区域可以分为两类,一类染色质的酶切信号随着酶切时间的增加而逐渐降低,而另一类随着酶切时间的增加其酶切信号会保持不变或者呈现逐渐升高的现象,通过对这两类染色质区域进行功能注释,发现第一类染色质多分布在活跃基因区域,而第二类染色质所分布的基因则多处于静息状态。在此基础上,研究者开发了一种定量染色质状态和基因表达潜力的方法。通过基因启动子区域染色质开放潜能参数(Chromatin Opening Potential Index,COPI),他们成功地对mES细胞中相关基因的转录潜能进行了评估。为了探索该方法的实用性,研究者验证了具有高COPI分值的基因,如Lfng、Ypel1、Scrt2和Ildr2等,这些基因的启动子处于部分开放状态,在mES细胞中不活跃,但在神经元祖细胞(NPC)分化过程中很容易被激活。其中,除了已经报道的参与NPC分化发育过程的基因Lfng和Scrt2外,更重要的是,通过COPI的预测,研究者发现Ildr2和Ypel1在小鼠NPC体外分化过程中也起着重要作用。因此,该研究能够通过对静息态染色质的结构进行分析从而预测基因转录潜能,为探索和发现多种生物过程的新的调控基因提供了一个强有力的工具,例如发育和分化过程中的关键调控因子,以及神经和免疫反应中的早期基因等。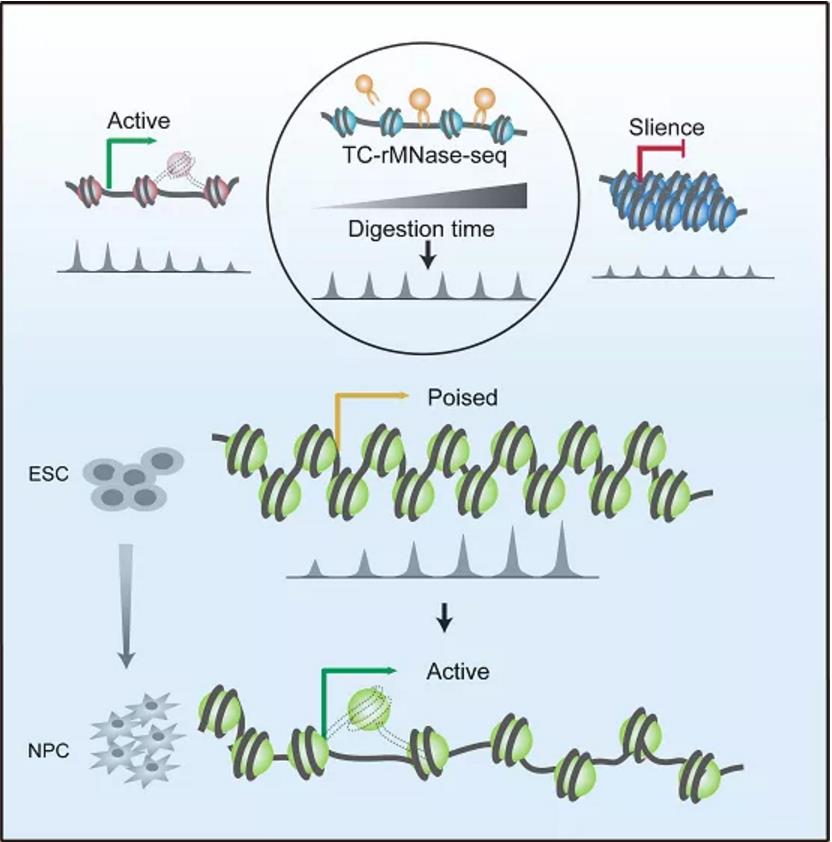
图示:通过鉴定局部染色质状态预测基因转录潜能的模型
中国科学院生物物理研究所李国红研究员和阮海荷博士以及暨南大学附属深圳市宝安区妇幼保健院许雪青研究员为该项工作的共同通讯作者,李国红组副研究员于娟、助理研究员熊朝阳以及许雪青组硕士研究生卓宝文(已毕业)为共同第一作者。该研究工作得到了国家自然科学基金、科技部重点研发计划、中国科学院先导专项、中国科学院前沿科学重点研究项目、HHMI国际研究学者以及深圳市卫计委医疗卫生"三名工程"项目的共同资助。
文章链接:https://www.sciencedirect.com/science/article/pii/S2211124720309347


