

Nature:Josefowicz/李海涛合作发现组蛋白H3.3磷酸化可增强刺激诱导基因的转录
日期:2020-07-23 浏览次数:13823
本文来源:BioArt
组蛋白是真核生物染色体的主要组成成分,不仅能保证DNA的正常复制,重组和修复,同时,组蛋白的翻译后修饰(post-translation modifications, PTMs)对基因转录有着重要的调控作用。
组蛋白H3有3个变体,分别是H3.1、H3.2和H3.3。组蛋白H3.3是一类保守的“祖先级”H3变体,是单细胞真核生物(如酵母)中组蛋白H3的唯一形式。与高等生物中复制依赖的组蛋白变体H3.1和H3.2不同,组蛋白变体H3.3主要参与染色质动态活动调控,在转录、DNA损伤修复和有丝分裂等过程中发挥着重要作用。组蛋白变体H3.3 与“经典”的组蛋白H3.1或H3.2的一个重要区别在于其氨基端第31位残基为丝氨酸(Ser或S), 而H3.1/2为丙氨酸(Ala或A)。这一氨基酸组成差异对于组蛋白H3.3的功能发挥有何意义是一个领域关注热点。
2014年,清华大学李海涛团队与美国MD Anderson癌症中心的石晓冰教授实验室曾在Nature发文,首次发现抑癌因子ZMYND11是H3.3变体第36位赖氨酸三甲基化(H3.3K36me3)的新型识别因子,并揭示其在抑制转录延伸过度活化方面的功能(差不多同一时期,哈佛大学施扬教授团队在Molecular Cell上也报道了类似的工作)。在这一过程中,丝氨酸31因为其有别于丙氨酸31的氢键形成能力,贡献了ZMYND11对H3.3的特异性识别【1】。H3.3的第31位丝氨酸还可被磷酸化修饰,然而目前关于H3.3S31和其磷酸化形式(H3.3S31ph)的功能和生物物理机制还需进一步研究【2-5】。
2020年7月22日,来自康奈尔大学的Steven Z. Josefowicz实验室、清华大学的李海涛教授团队以及洛克菲勒大学David Allis实验室等合作在Nature发表研究 Histone H3.3 phosphorylation amplifies stimulation-induced transcription,该研究阐明了H3.3S31ph通过招募和拮抗不同的染色质调控因子从而增强刺激诱导的基因转录的机制。
复杂生物体可快速诱导特定基因的转录以响应各种环境变化,然而目前还不清楚这些刺激诱导的基因(stimulation-induced genes)是如何高效地被转录机器识别并激活的。特别地,这些基因的调控发生在由组蛋白和DNA凝集形成的染色体中,因此研究者猜测刺激诱导的转录可能受染色质调节机制和信号激活因子的共同作用。
为了鉴定可能参与刺激响应过程的染色质调节因子,研究者纯化了用细菌脂多糖(lipopolysaccharide,LPS)处理的小鼠骨髓来源的巨噬细胞(bone marrow-derived macrophages ,BMDMs)的组蛋白,并利用质谱鉴定组蛋白修饰的变化。特别地,H3.3S31ph由静息状态的基本无法检测变成受刺激后显著上升。进一步研究显示H3.3S31ph主要位于受LPS诱导激活的基因的gene body区,且是由磷酸化酶IKKα通过结合RNA聚合酶II(pol II)以共转录的形式催化的,这与前人的研究结果一致【6】。
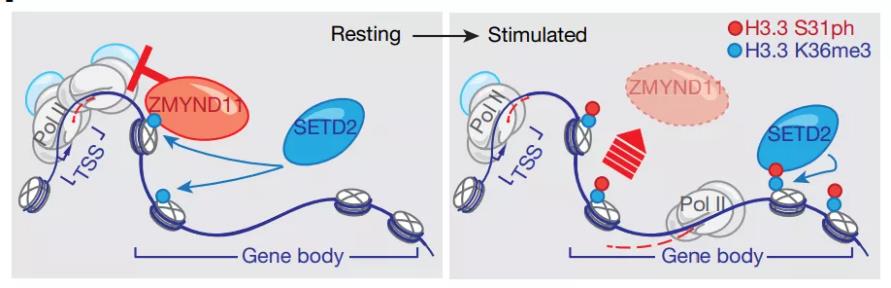
那么,位于诱导激活基因的H3.3S31ph的功能是什么?H3.3S31ph如何影响这些基因的转录呢?研究者注意到H3K36me3修饰在刺激诱导型基因上的定位与H3.3S31ph高度重合。在高等动物中,只有SETD2甲基转移酶能够完成H3K36位点的三甲基化, 而其他同源酶例如NSD2只能完成一甲基化/二甲基化。李海涛团队曾在2016年首次解析出SETD2结合组蛋白底物的复合物结构【7】。结构分析表明,发现H3S31位点位于一个正电荷富集区,这对于结合带有负电荷的H3.3S31ph修饰极为有利。通过体外酶活实验,作者发现H3.3S31的磷酸化修饰能够提高SETD2的甲基转移活力,对NSD2则没有促进作用。进一步的复合物结构解析证明H3.3S31ph的磷酸基团与SETD2的两个碱性残基K1600和K1673存在静电吸引和水介导的氢键作用,进而揭示了H3.3S31ph促进SETD2酶活的分子基础。有意思的是,序列分析表明K1600和K1673残基只在SETD2中存在且相当保守,而在NSD2等其他酶中则被替换为酸性或极性氨基酸,这也就完美解释了为什么H3.3S31ph不能促进与NSD2直接相关的组蛋白H3.3K36me2的修饰水平。总之,生化与结构实验共同揭示出H3.3S31ph促进H3.3K36me3的顺式激活是一种组蛋白修饰会话(cross-talk)机制。
在H3的尾巴上引入S31ph是否还会影响其他染色质调节因子对H3的结合呢?S31ph可能会促进某些蛋白对H3的结合,比如前述的SETD2;也可能抑制某些蛋白与H3结合。已知肿瘤抑制因子ZMYND11能够特异性地识别H3.3K36me3修饰,抑制转录延伸的过度活化【1】。研究者通过ZMYND11的ChIP-seq分析, 结合结构分析和ITC滴定实验,发现H3.3S31位点产生磷酸化后会因为体积排斥效应,“踢走”原本结合的ZMYND11,从而解除转录延伸共抑制子ZMYND11的抑制效应,促进了H3.3S31ph靶基因的活跃转录。
总的来说,该研究发现H3.3S31ph通过促进SETD2催化H3K36me3和拮抗ZMYND11对H3K36me3的阅读大幅度地促进刺激诱导基因的转录(图1)。值得注意的是,该研究的蛋白结构解析为阐明不同表观遗传修饰间的交流(cross talk)提供了直接且有力的证据。快速响应外界刺激并启动特定基因转录的过程与多种细胞反应和疾病相关,该特异性转录激活过程可能比通用转录机制更适合作为疾病治疗的靶点,这项研究发现组蛋白磷酸化修饰正是开启这一过程的关键,具有重要意义。
据悉,清华大学医学院李海涛教授与美国康奈尔医学院Steven Josefowicz助理教授是本论文的共同通讯作者,洛克菲勒大学C David Allis教授实验室博士后Anja Armache博士和李海涛教授实验室已毕业研究生杨爽博士为本论文共同第一作者,李海涛教授实验室岳媛博士参加了本项工作。
参考文献:
1. Wen, H. et al. ZMYND11 links histone H3.3K36me3 to transcription elongation and tumour suppression. Nature 508, 263–268 (2014)
2. Filipescu, D., Szenker, E. & Almouzni, G. Developmental roles of histone H3 variants and their chaperones. Trends Genet. 29, 630–640 (2013)
3. Henikoff, S. & Smith, M. M. Histone variants and epigenetics. Cold Spring Harb. Perspect. Biol. 7, a019364 (2015)
4. Kamada, R. et al. Interferon stimulation creates chromatin marks and establishes transcriptional memory. Proc. Natl Acad. Sci. USA 115, E9162–E9171 (2018).
5. Shi, L., Wen, H. & Shi, X. The histone variant H3.3 in transcriptional regulation and human disease. J. Mol. Biol. 429, 1934–1945 (2017).
6. Thorne, J. L., Ouboussad, L. & Lefevre, P. F. Heterochromatin protein 1 gamma and IκB kinase alpha interdependence during tumour necrosis factor gene transcription elongation in activated macrophages. Nucleic Acids Res. 40, 7676–7689 (2012)
7. Shuang Yang , Xiangdong Zheng , Chao Lu , Guo-Min Li , C David Allis , Haitao Li, Molecular basis for oncohistone H3 recognition by SETD2 methyltransferase, Genes Dev 2016 Jul 15;30(14):1611-6.doi: 10.1101/gad.284323.116.