雷鸣组揭示CST复合物调控端粒复制和确保端粒末端稳定性的分子机制
日期:2020-07-15 浏览次数:10930
本文来源:BioArt
真核生物线性染色体必须克服末端保护和末端复制问题。端粒是位于真核细胞线性染色体DNA的末端,由串联重复的DNA序列与端粒结合蛋白组成的特殊结构。端粒结构的存在很好的解决了染色体末端保护和复制问题,对确保基因组的稳定性和完整性具有极其重要的作用。端粒的长度主要由一个特化的反转录酶—端粒酶—维持,利用其自身RNA亚基的一部分为模板增加端粒末端DNA重复序列。然而,人的正常成年体细胞内没有端粒酶的活性,端粒会随着细胞分裂增加而逐渐缩短。一旦端粒消耗殆尽,染色体的稳定性被打破,就会起始细胞衰老和凋亡。许多与衰老相关的疾病以及早衰综合症都与端粒的加速缩短有关,因此端粒的缩短也被视为引起机体衰老的原因之一。在大约85%的肿瘤细胞中检测出端粒酶活性,而且相关研究表明失活端粒酶可以抑制肿瘤细胞端粒的延伸,从而抑制癌细胞的增殖。因此,通过抑制端粒酶活性来阻止癌细胞的生长成为治疗癌症的又一强有力的方法,成为国内外研究的热点。CST复合物是真核生物中高度保守的端粒结合蛋白复合物,在芽殖酵母中有Cdc13-Stn1-Ten1三个蛋白质组成。CST复合物在结构上被认为是端粒特异的单链DNA结合RPA复合物,但体外生化数据表明CST和RPA复合物各亚基的化学计量数并不是一致的【1,2】。因此,CST复合物整体结构信息的解析对回答CST和RPA复合物的关系至关重要。CST复合物在端粒维持和端粒末端保护中具有重要的功能,比如Cdc13通过协调Stn1和端粒酶亚基Est1的相互作用维持端粒复制和端粒保护的动态平衡。2018年1月,雷鸣课题组解析了Cdc13与Est1相互作用的结构结构信息,并揭示了端粒酶招募至端粒的分子机制【3】。因此,进一步解析Cdc13与Stn1相互作用的结构信息对于揭示CST复合物调控端粒复制的分子机制具有重要的意义。
2020年7月13日,上海交通大学医学院附属第九人民医院精准医学研究院雷鸣课题组在Nature Structural & Molecular Biology在线发表了题为“Structural insights into telomere protection and homeostasis regulation by yeast CST Complex”的研究论文,该工作从结构层面阐明了芽殖酵母中CST复合物在端粒上的组装方式,并揭示了CST复合物调控端粒复制和确保端粒末端稳定性的分子机制。
该研究工作分别解析了Cdc13-ssDNA,Cdc13-Stn1和Stn1-Ten1相互作用的晶体结构,并重构了CST复合物组装的结构模型。该工作表明Cdc13和Stn1形成一个异源四聚体的环形结构,进一步结构模型搭建表明CST复合物以该异源四聚体为核心,形成三个亚基化学计量数为2: 2: 2的高级结构。而RPA复合物为1: 1: 1,表明CST复合物并非之前公认的端粒特异RPA复合物。在这篇文章在线前不久,Thomas R. Cech课题组报道了人源CST(CTC1-STN1-TEN1)结合端粒ssDNA的电镜结构【4】。结合2015年11月雷鸣课题组发表的四膜虫中独特CST复合物p75–p45–p19的结构信息【5】,不同物种的CST复合物结构比较表明不同物种中CST复合物高级结构差异很大:芽殖酵母中CST复合物为2: 2: 2的六聚体组装方式;人源CST复合物是以十二聚体的组装方式结合端粒ssDNA;四膜虫中CST复合物p75–p45–p19以1: 1: 1锚链在端粒酶上。基于结构信息,研究人员从功能上阐明了Cdc13-Stn1相互作用竞争端粒酶在端粒的结合,从而抑制端粒的过度复制。另外也研究了CST复合物中Stn1-Ten1相互作用在端粒末端保护中的关键作用。最重要的是,雷鸣课题组连续两篇重要研究成果分别揭示了Cdc13-Est1相互作用招募端粒酶至端粒和激活端粒酶活性,以及CST复合物终止端粒酶活性防止端粒过度复制的分子机制,描绘了CST复合物在端粒维持中功能的分子模型(图1)。
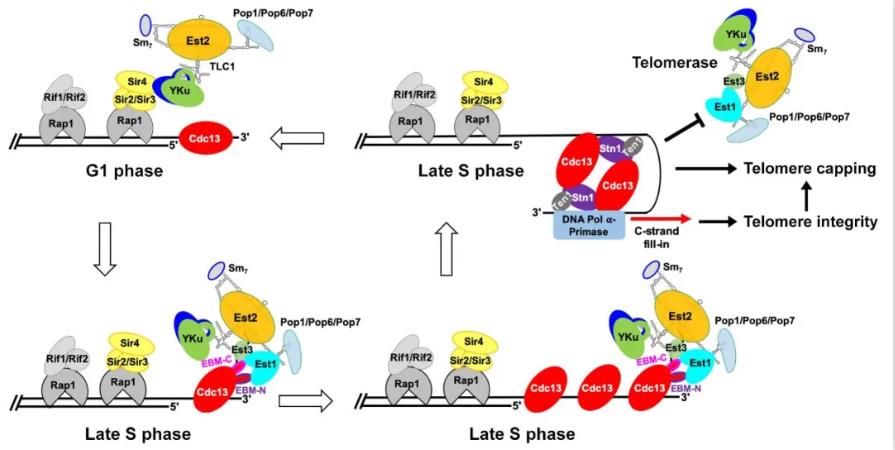
图1. CST复合物调控端粒复制和确保端粒末端稳定性的分子机制
总的来说,该工作结合结构生物学和遗传学技术手段,不仅揭示了CST复合物调控端粒复制和确保端粒末端保护的分子机制,也为探究CST复合物和端粒酶相关疾病的分子机制和研发相关药物提供重要的理论依据。
原文链接:
https://doi.org/10.1038/s41594-020-0459-8
参考文献:
1.Gelinas, A.D. et al. Telomere capping proteins are structurally related to RPA with an additional telomere-specific domain. Proc Natl Acad Sci USA 106, 19298-303, doi: 10.1073/pnas.0909203106 (2009).2.Lue, N.F. et al. The telomere capping complex CST has an unusual stoichiometry, makes multipartite interaction with G-Tails, and unfolds higher-order G-tail structures. PLoS Genet 9, e1003145, doi: 10.1371/journal.pgen.1003145 (2013).3.Chen, H. et al. Structural Insights into Yeast Telomerase Recruitment to Telomeres. Cell 172, 331-343, doi: 10.1016/j.cell.2017.12.008 (2018).4.Lim, C.J. et al. The structure of human CST reveals a decameric assembly bound to telomeric DNA. Science 368, 1081-1085, doi: 10.1126/science.aaz9649 (2020).5.Wan, B. et al. The Tetrahymena telomerase p75-p45-p19 subcomplex is a unique CST complex. Nat Struct Mol Biol 22, 1023-6, doi: 10.1038/nsmb.3126 (2015).


