Cell | 向烨/章新政合作解析虫媒病毒跨物种传播分子机制
日期:2023-04-25 浏览次数:20132
来源:BioArt
虫媒病毒可以通过许多种昆虫媒介来传播,其中蚊子和蜱是最常见的传播病毒的昆虫媒介【1】。在热带和亚热带地区,登革热病毒(DENV)、黄热病毒(YFV)、基孔肯雅病毒(CHIKV)等虫媒病毒对当地居民的健康造成了极大的威胁。根据世界卫生组织的数据,每年有超过4亿人感染登革热病毒,50万人发生严重的登革热,其中约有2.5%至5%病死率【2】。黄热病对热带和亚热带地区的原住民和旅游者造成了严重的健康威胁。据世界卫生组织的数据,每年有超过200,000例黄热病,其中90%以上发生在非洲,每年导致30,000至60,000人死亡。基孔肯雅病毒感染通常会导致发热、关节疼痛和皮疹等症状,部分感染者会有长时间的关节炎和疼痛。根据世界卫生组织(WHO)的报道,自2004年以来,基孔肯雅病毒感染的发病率不断上升,尤其在2013年至2015年,该病毒在加勒比地区和拉丁美洲大规模爆发,据估计超过140万人受感染。虫媒病毒引起的传染病在我国南方地区也频繁发生。自2018年以来,我国多个省份陆续受到登革热疫情的威胁,其中广东和贵州等地的疫情较为严重。登革热病毒(DENV)、黄热病毒(YFV)、基孔肯雅病毒(CHIKV)分别属于黄热病毒属及甲病毒属,都是具有正链RNA基因组的囊膜病毒【3】。对于这些虫媒病毒传播及致病分子机制的研究对于相关药物及疫苗的开发具有重要意义。
虫媒病毒能感染从无脊椎动物如昆虫,到低等脊椎动物如鱼类,到哺乳动物如马,人等多种物种,其宿主物种跨越巨大【4】。虫媒病毒通过两种可能途径实现跨度极大物种之间传播,一种是识别不同宿主细胞上的不同受体,另一种是通过识别不同宿主细胞上的通用受体。目前已知介导甲病毒感染的受体MXRA8以及LDLRAD3并不广泛保守的存在于不同物种中,特别是跨度较大的物种【5,6】。介导病毒跨越差异极大的不同物种之间传播的通用受体鲜有报道,病毒与这类受体的结合模式及其介导的跨物种传播机制尚不清楚。2021年的一篇研究报道表明载脂蛋白受体VLDLR(very-low-density lipoprotein receptor)是多种甲病毒,包括塞姆利基森林病毒(Semliki Forest Virus, SFV),感染蚊虫、鼠、马、人等多个物种的通用受体【7】。
2022年春节刚过,清华大学向烨和中科院生物物理所章新政两个研究组合作开始研究SFV与受体的结合模式及高分辨结构,并于2023年4月24日,在 Cell 杂志在线发表了该研究成果,题目为Structure of Semliki Forest virus in complex with its receptor VLDLR。
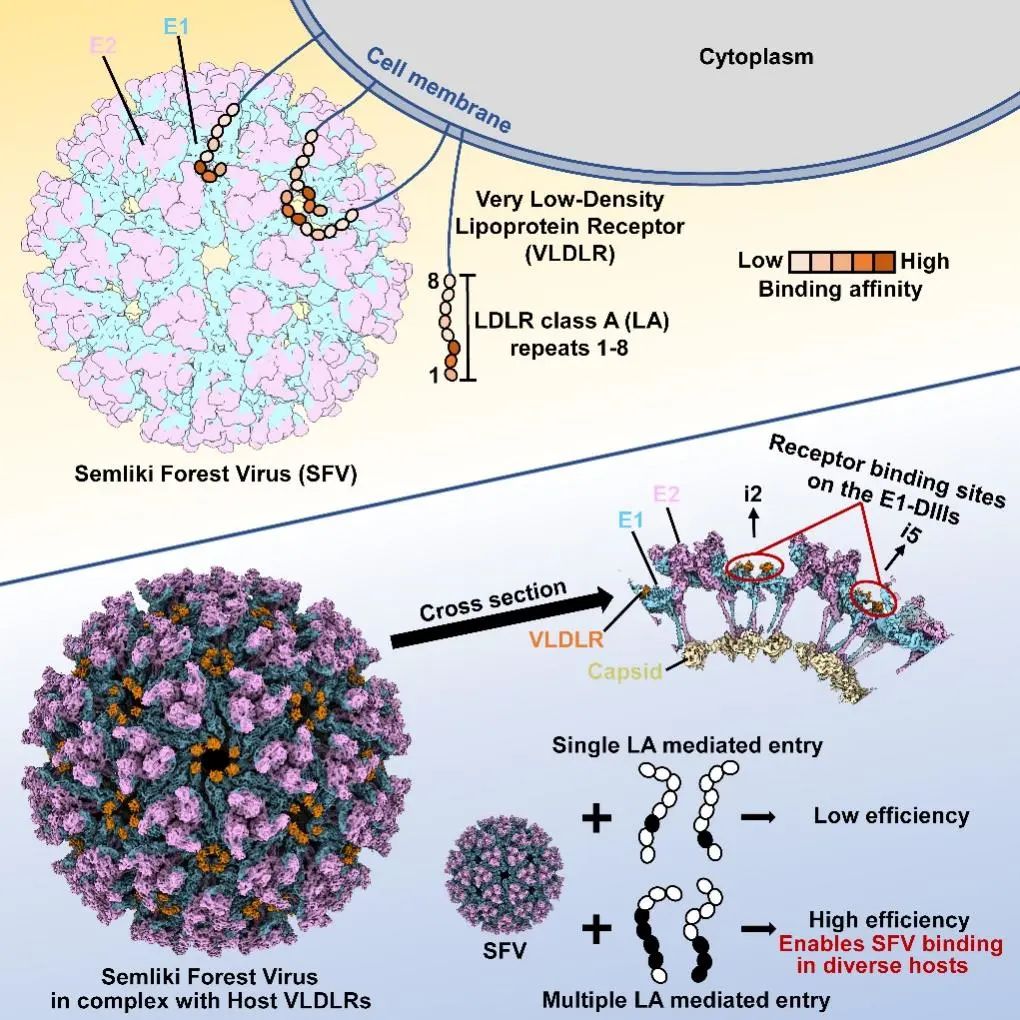
研究团队首先建立了SFV VLP的表达系统,并进一步制备了SFV VLP与VLDLR胞外域的复合物。随后,研究团队收集了冷冻电镜数据并解析了结构。已有研究普遍认为位于Spike近囊膜区域的E1主要负责病毒与宿主的膜融合,而位于病毒远离囊膜区域spike上的E2参与受体结合【8】。LDLRAD3和MXRA8都结合在病毒spike(刺突)上相邻E1-E2二聚体形成的狭缝中,病毒的E1、E2蛋白均参与了与受体的结合【9-12】。而此次结构分析发现VLDLR结合在SFV E1蛋白贴近病毒囊膜区域的DⅢ结构域(E1-DⅢ)上,这一结合位点完全不同于另外两种甲病毒受体MXRA8及LDLARAD3的结合位点(位于远离病毒囊膜的刺突上),且 SFV E2并没有参与和受体VLDLR的结合。这种结合方式刷新了关于甲病毒与受体相互作用的认知。然而,所解析的复合物结构虽然总体分辨率达到3.0埃,但受体部分密度并不能提供任何细节。造成这一结果的可能原因是受体并非以一种固定模式结合于SFV VLP。研究团队进一步发现VLDLR通过其胞外多个富含半胱氨酸重复序列结构域(LDLR class A repeats,LAs)与SFV E1-DⅢ结合,而新鉴定的受体结合位点在病毒表面的特殊分布使得串联的多个LA结构域和相邻的受体结合位点的同时结合成为可能。
VLDLR胞外有8个相似的LA结构域,但通过结构无法区分是哪几个LA结构域与SFV VLP结合。于是研究团队把8个LA结构域分别拆分出来,构建质粒并表达纯化所有单个LA结构域,同时利用BLI测定其与SFV E1-DIII的结合解离常数。最终发现第三个LA(LA3)与SFV E1-DIII结合最紧密,但其解离常数也只是在1 M左右。其它单个LA与SFV E1-DIII的结合力也都不强。为解析LA与SFV的详细相互作用,研究团队随即选择LA3与SFV VLP形成复合物并解析结构。LA3与SFV VLP复合物结构中在2次轴附近结合的LA3密度清晰可见,其中绝大部分残基密度可见并可以依据其建立模型。基于LA3与SFV VLP复合物结构,LA与SFV E1-DIII作用的细节被清晰解析。与MXRA8及LDLRAD3相比,LA3与E1-DIII的结合面要小得多,只有前两者的1/3-1/2。这也解释了其相对较弱的亲和力。LA3-SFV VLP复合物结构的测定进一步说明VLDLR胞外多个串联LA结合SFV的模式不固定,造成不同LA结构域密度之间被平均,从而细节无法被解析。有趣的是结合5次轴附近E1-DIII上的LA3密度很差,进一步分析表明由于有严重的空间位阻,以LA3的结合方式不能同时占有相邻的两个E1-DIII位点,因此5次轴附近LA3的占有率大大低于2次轴附近的LA3.
单个LA结构域与病毒的结合力弱,其解离常数为1-30 M,且通过单个LA结构域难以介导病毒的入侵;而多个串联LA结构域与病毒的结合显著增强,其解离常数为1-100 nM,且能高度有效的介导病毒入侵。SFV表面受体结合位置E1-DⅢ在二次轴、五次轴附近相互靠近且密集排布,为多个LA结构域可能的协同结合提供了先决条件。通过解析SFV VLP与多个不同串联LA复合物结构,研究团队发现多个串联LA可同时结合SFV VLP多个E1-DIII位点,且并以一种非固定的模式结合(图1),且不同串联LA在各个E1-DIII位点有不同占有率。但总体规律是串联的LA数目越多,占有率越高。在五次轴附近,不同串联LA复合物显示类似的结合方式,与LA3的结合方式相比,串联LA中与E1-DIII结合的LA结构域都发生约16度的旋转以避免各自之间产生空间位阻,以及利于协同结合E1-DⅢ。与单个LA结合时相比,串联形式中的LA虽然在转动过程中大部分与E1-DⅢ的相互作用会被破坏,但是一些关键位点上的相互作用依旧保留着,而且通过LA之间的协同作用使得多个串联LA可以同时结合E1-DIII,弥补了单个LA上非关键性相互作用的损失,大大增强了病毒与受体的结合力。从而,这种多个串联LA协同作用模式能够增加受体对单个LA中结合能力丢失的容忍度。不同物种VLDLR大多数LA序列虽然差异较大,但与病毒作用关键残基W132、E137和D139完全保守。VLDLR LA结构域独特的协同结合模式,使其在介导病毒感染时能够耐受来自不同进化层次物种间序列的差异,实现跨度很大的多物种之间的病毒传播。
综上,通过结构生物学手段,结合多种生化方法,向烨与章新政团队合作阐述了不同宿主中进化上相对保守的通用细胞受体通过其多个相似结构域弱相互作用的协同工作与病毒特异性结合,从而介导病毒跨物种传播。
清华大学医学院向烨研究员和中国科学院生物物理研究所章新政研究员为该论文的共同通讯作者,章新政课题组副研究员曹端方、向烨课题组博士后马丙婷和章新政课题组博士研究生曹紫怡为该论文的共同第一作者。
回顾两个合作团队在甲病毒研究中的历史,2010年当时还在Purdue大学Michael G. Rossmann研究组的博士后孙思洋、向烨、章新政合作研究囊膜虫媒甲病毒基孔肯雅病毒(CHIKV)的高分辨结构,最终获得基孔肯雅病毒5.5埃分辨率结构【13】。在基孔肯雅病毒结构研究过程中,发现很难得到此类囊膜病毒的近原子分辨率结构,可能原因是病毒的外层E蛋白具有一定的形变导致其结构不具有完美的二十面体对称性。2018年章新政研究组在同属甲病毒的囊膜病毒SINV上运用其研究组发展的block based 算法,解决了此类病毒外层E蛋白形变问题,获得3.5埃近原子分辨率结构,并发现脂质分子参与SINV E蛋白组装【14,15】。2019年在导师Michael G. Rossmann去世后,向烨研究组开始研究虫媒囊膜病毒,并建立了甲病毒病毒样颗粒组装系统,时隔9年后与章新政组合作重启甲病毒CHIKV结构及其与受体MXRA8复合物结构研究,并很快得到总体分辨率为3.0埃复合物结构,但由于受体分子部分密度不足以支持准确建立模型未发表相关结果,而同一时间,另两个研究组通过X射线晶体学获得结构并发表在Cell杂志上【11,12】。
2021年甲病毒VEEV的受体分子LDLRAD3被报道后,向烨与章新政两个研究组再次合作研究甲病毒VEEV与受体分子LDLRAD3结构,发现LDLRAD3特异性结合VEEV的结构基础,同时通过VEEV高分辨结构解析了其E蛋白与囊膜内层capsid蛋白的详细相互作用【9】,矫正了之前基于低分辩结构的一些错误认识。
新型受体通过多个LA串联结构域协同介导甲病毒SFV的跨物种传播(图片来自Cell文章graphic abstract)
原文链接:https://doi.org/10.1016/j.cell.2023.03.032
参考文献: