哈尔滨工业大学黄志伟团队首次解析CRISPR-Cas III-E型效应器与TPR-CHAT的结构
日期:2022-10-27 浏览次数:23568
来源:iNature
原核生物和病毒已经进行了数十亿年的进化斗争。细菌和古生菌使用聚集的有规律的间隔短回文重复序列(clustered regularly interspaced short palindromic repeats, CRISPR)-CRISPR相关(Cas)适应性免疫系统来防止病毒感染。
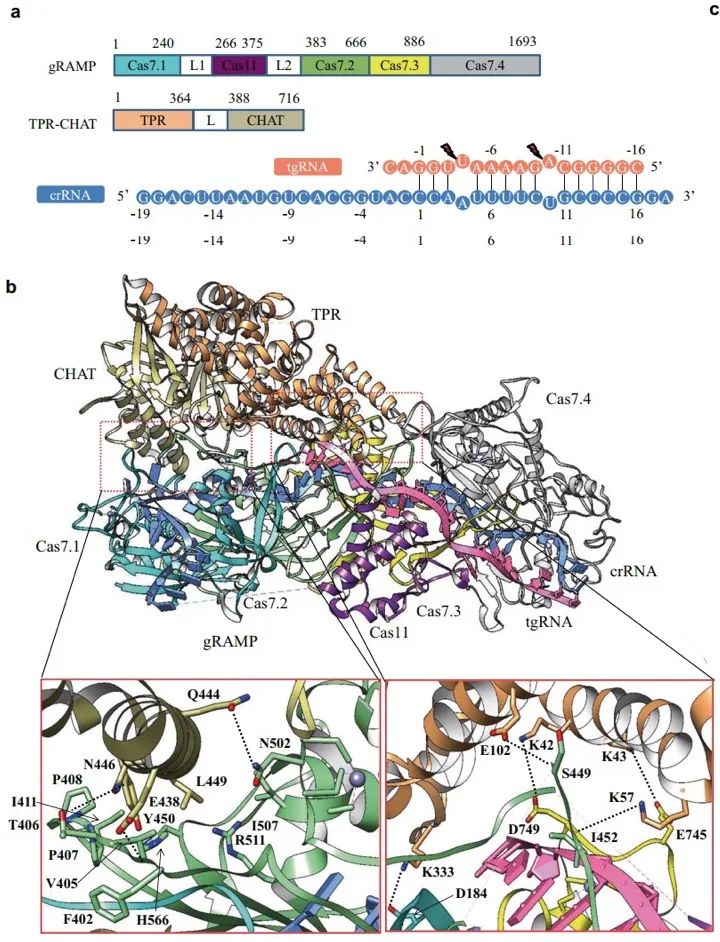
与传统III型CRISPR-Cas系统的效应子不同,gRAMP是一个由4个Cas7蛋白和1个Cas11蛋白融合在一起的单一效应子蛋白。由于其独特的结构和特定的切割活性,gRAMP有望被设计成一种强大的RNA编辑工具。有趣的是,编码caspase样肽酶TPR-CHAT的基因经常与gRAMP基因簇同时出现。TPR-CHAT与gRAMP直接相互作用,表明CRISPR-Cas系统与caspase肽酶之间存在功能关系。然而,念珠菌“Scalindua brodae”(Sb-gRAMP)中,gRAMP识别和切割目标ssRNA的确切机制,以及CRISPR引导的caspase复合物的分子结构尚不清楚。2022年10月24日,哈尔滨工业大学黄志伟团队在Cell Research 杂志在线发表题为“Cryo-EM structure of the type III-E CRISPR-Cas effector gRAMP in complex with TPR-CHAT”的研究论文,该研究报告了CRISPR III-E型效应器与TPR-CHAT结合的结构,为阐明CRISPR- Cas系统与caspase肽酶之间的功能关系提供了重要线索。CRISPR-Cas基因座由两部分组成:CRISPR和Cas基因。根据最新的系统发育分类,CRISPR-Cas系统可分为两类,再细分为六类。第1类CRISPR-Cas系统以多蛋白效应器为特征,由I型、III型和IV型CRISPR-Cas系统组成。第2类CRISPR-Cas系统以单个核酸酶蛋白为特征,由II型、V型和VI型CRISPR-Cas系统组成。II型Cas9和V型Cas12系统已被设计成基因组编辑工具,并已成功应用于广泛的生物体中。与Cas9相比,VI型Cas13系统具有RNA引导的RNase活性和非特异性切割的特性。最近,CRISPR-Cas的一种新的亚型,III-E型被鉴定出来并命名为gRAMP7或Cas7-11。
在这项研究中,研究人员测定了III-E型效应器Sb-gRAMP–crRNA与TPR-CHAT复合物有和没有靶ssRNA的结构的冷冻电子显微镜(cryo-electron microscopy, cryo-EM)结构,分辨率分别为3.0 Å和2.9 Å。得到的图谱帮助构建gRAMP–crRNA–ssRNA–TPR-CHAT复合物的全新模型。结构表明,gRAMP - crRNA - ssRNA - TPR-CHAT复合体的整体结构采用“L”形构象,由一个gRAMP、一个TPR-CHAT、一个37-nt crRNA和一个18-nt靶ssRNA组成。
gRAMP与TPR-CHAT复合物的Cryo-EM结构(图源自Cell Research )
进一步研究表明,gRAMP - crRNA - ssRNA - TPR-CHAT的长度和宽度分别约为145 Å和96 Å。gRAMP由一个Cas11域和四个Cas7域组成。4个Cas7结构域(Cas7.1-Cas7.4)沿crRNA一侧堆叠,形成细丝。在gRAMP中的Cas7结构域显示出类似于III-A型Csm3和III-B型Cmr4中观察到的RRM折叠。gRAMP和Csm3/Cmr4中Cas7结构域的一个显著区别是每个Cas7结构域都包含一个锌指基序。在Cas7.1, Cas7.2和Cas7.4结构域,锌离子由C2C2锌指基序配位。相比之下,Cas7.3结构域的锌离子是由CCCH锌指基序配位的。Cas11结构域位于crRNA的另一侧,与Cas7.2、Cas7.3和靶ssRNA相互作用。与III-A型Csm2和III-B型Cmr5结构相似,在gRAMP中Cas11结构域呈现螺旋束构象,一侧有3个α-螺旋,另一侧有2个α-螺旋。III-E型效应器系统的结构与III-A型和III-B型效应器系统明显不同。Cmr/Csm综合体的整体结构呈现出胶囊状的架构。4个Cmr4(3个Csm3)和3个Cmr5(2个Csm2)亚基构成双螺旋骨干,一端(头部)由Cmr1-Cmr6 (Csm5)覆盖,另一端(尾部)由Cmr2-Cmr3 (Csm1-Csm4)覆盖。这些结果提供了Caspase体系结构的原子视图。结合诱变实验和结构信息,可以进一步确定靶ssRNA裂解的gRAMP中的两个催化残基。由于结构的灵活性,研究人员没有观察到插入域(1031-1390残基)和部分crRNA(碱基19-38)。综上所述,这项研究揭示了gRAMP识别crRNA和靶ssRNA的机制。该结构为crRNA前处理和靶ssRNA切割提供了新的见解。原文链接: https://www.nature.com/articles/s41422-022-00738-3


