

Cell:张宏团队揭示内质网表面钙瞬变决定自噬体在内质网形成
日期:2022-10-08 浏览次数:7382
来源:BioArt
研究者首先发现钙离子快速螯合剂BAPTA-AM可以抑制参与自噬起始的FIP200复合物在内质网上形成凝聚体,但这一过程不能被慢速钙离子螯合剂EGTA-AM阻断。这提示快速的局部的钙离子变化,而非稳态的钙离子浓度变化,可能参与了自噬起始过程。研究人员构建了内质网跨膜结构域CYB5与快速钙离子探针GCaMP6f的融合蛋白,将GCaMP6f定位于内质网外膜表面朝向胞浆侧,以检测内质网外膜表面钙离子浓度的变化。利用多模态超分辨活细胞成像技术(Multi-SIM),研究者发现在饥饿或Torin1处理等自噬诱导条件下,内质网表面发生钙瞬变/钙振荡,且这些钙信号能被BAPTA-AM阻断(Figure 1)。
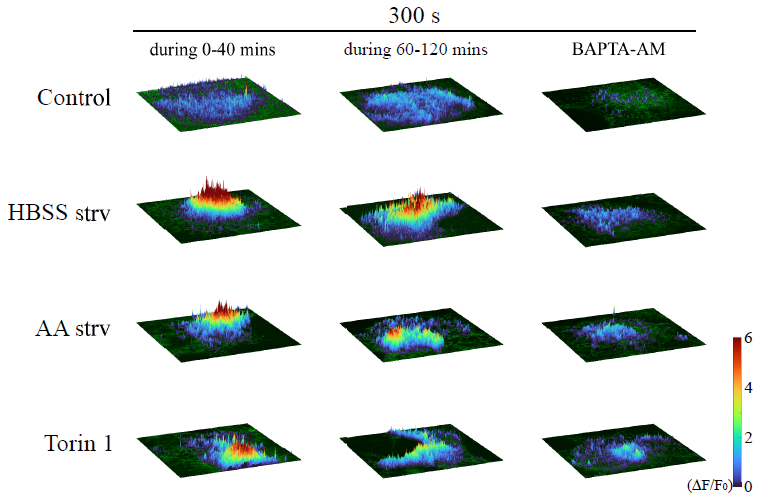
Figure 1. Multi-SIM分析不同刺激条件下钙离子浓度变化的3D曲面图。
研究发现,自噬诱导条件下,内质网表面发生的钙瞬变/钙振荡触发FIP200复合物发生液-液相分离,形成具有高度动态且易于融合的液滴状FIP200凝聚体(Figure 2)。FIP200凝聚体通过与内质网膜蛋白VAPs和ATLs结合,稳定定位于内质网上,并在内质网上移动,与其他的FIP200凝聚体融合。在达到一定大小后,FIP200凝聚体停止融合,成为内质网上的自噬体起始位点,招募下游自噬蛋白,启动自噬体的形成。研究者还发现ATG9囊泡参与调节FIP200复合物的相分离,并调节FIP200凝聚体在内质网上的空间构建。
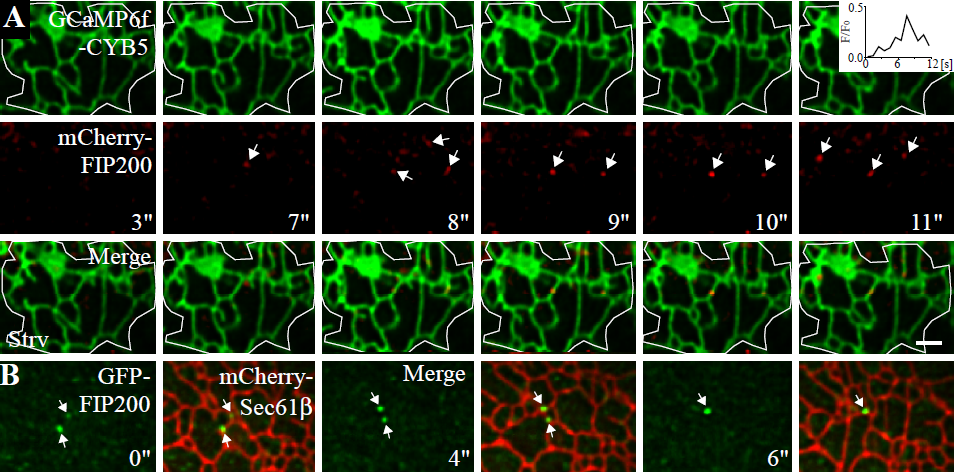
Figure 2. (A) 内质网外膜钙瞬变触发FIP200复合物发生液-液相分离。(B) 内质网表面的FIP200凝聚体发生融合。
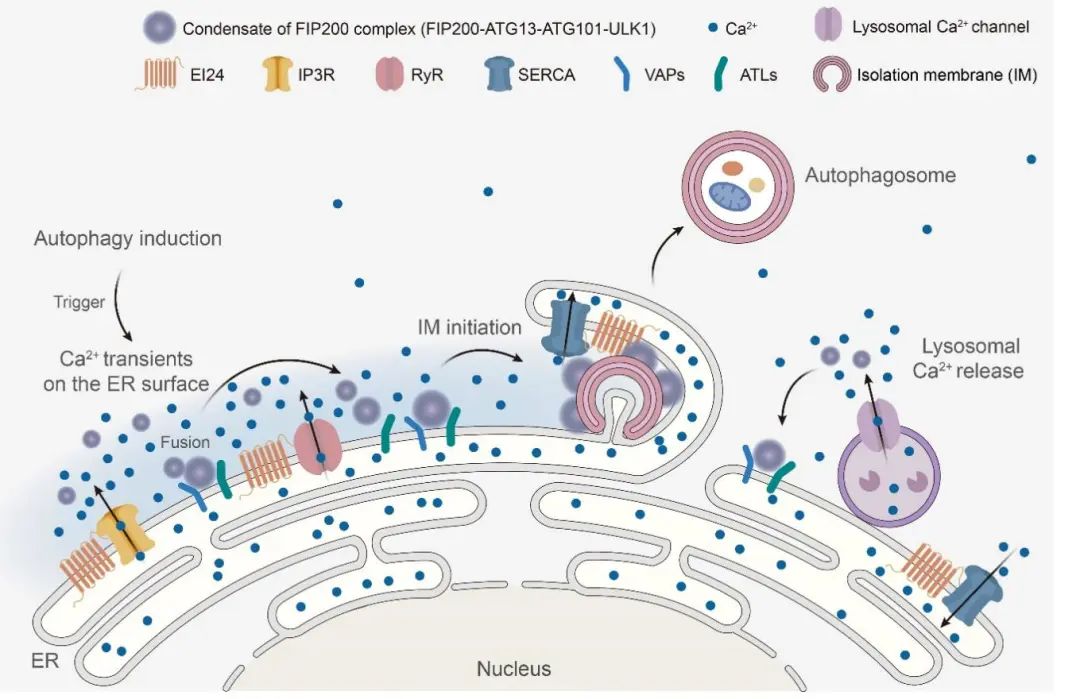
Figure 3. 内质网外膜钙瞬变触发FIP200复合物发生液-液相分离,并定位于内质网形成自噬起始位点的模式图。
1. Lamb, C.A., Yoshimori, T., and Tooze, S.A. (2013). The autophagosome: origins unknown, biogenesis complex. Nat. Rev. Mol. Cell Biol. 14, 759–774.
2. Zhao, Y.G., Codogno, P., and Zhang, H. (2021). Machinery, regulation and patho-physiological implications of autophagosome maturation. Nat. Rev. Mol. Cell Biol. 22, 733–750.
3. Nakatogawa, H. (2020). Mechanisms governing autophagosome biogenesis. Nat. Rev. Mol. Cell Biol. 21, 439-58.
4. Fujioka, Y., Alam, J.M., Noshiro, D., Mouri, K., Ando, T., Okada, Y., May, A.I., Knorr, R.L., Suzuki, K., Ohsumi, Y., et al. (2020). Phase separation organizes the site of autophagosome formation. Nature 578, 301-305.
5. Zhao, Y.G., and Zhang, H. (2018). Formation and maturation of autophagosomes in higher eukaryotes: a social network. Curr. Opin. Cell Biol. 53, 29–36.
6. Karanasios, E., Walker, S.A., Okkenhaug, H., Manifava, M., Hummel, E., Zimmermann, H., Ahmed, Q., Domart, M.C., Collinson, L., and Ktistakis, N.T. (2016). Autophagy initiation by ULK complex assembly on ER tubulovesicular regions marked by ATG9 vesicles. Nat. Commun. 7, 12420.
7. Tian, Y., Li, Z., Hu, W., Ren, H., Tian, E., Zhao, Y., Lu, Q., Huang, X., Yang, P., Li, X., et al. (2010). C. elegans screen identifies autophagy genes specific to multicellular organisms. Cell 141, 1042–1055.
8. Zhao, Y.G., Chen, Y., Miao, G., Zhao, H., Qu, W., Li, D., Wang, Z., Liu, N., Li, L., Chen, S., et al. (2017). The ER-localized transmembrane protein EPG-3/VMP1 regulates SERCA activity to control ER-isolation membrane contacts for autophagosome formation. Mol. Cell 67, 974-989.