

陈雷研究组报道血管扩张剂在KATP通道SUR2亚基上的结合位点
日期:2022-05-17 浏览次数:3797
供稿:陈雷研究组
ATP敏感钾离子通道(KATP)是由Kir6亚基和SUR亚基组成的异源八聚体,可被细胞内ATP抑制,Mg-ADP激活1。KATP通道在脑、胰岛和肌肉等组织中广泛表达,通过感受细胞代谢水平调节生理功能。KATP通道基因突变可导致新生儿糖尿病、先天性高胰岛素血症2、扩张性型心肌病3和坎图综合征4,5等一系列疾病,以KATP通道为靶点的多种药物小分子已批准应用于临床,用于治疗疾病。
KATP开放剂(KCO)是一类在Mg-ATP或Mg-ADP存在下可激活KATP通道的小分子。KATP通道的开放使质膜超极化,降低细胞的兴奋性,并抑制细胞内钙信号传导6。根据SUR亚基选择性,KCO可分为三类:SUR1特异性开放剂,如NN414(替芬那嗪);SUR2特异性开放剂,如 P1075 和 levcromakalim (Lev);以及非选择性开放剂,如二氮嗪。二氮嗪能激活胰岛KATP通道,可用于阻断胰岛素分泌,提高血糖水平以治疗低血糖症。SUR2特异性开放剂可松弛血管平滑肌,也被称为“血管扩张剂”,在临床中应用于治疗高血压、心绞痛和心律失常等心血管疾病6。这些药物兼有促进毛发生长的作用,广泛地用于治疗脱发,如OTC米诺地尔等。此外,SUR2特异性开放剂在其他组织疾病的临床治疗应用中也显示出前景,包括支气管扩张、膀胱松弛、癫痫和青光眼等7。然而,这些血管扩张剂是如何结合并激活KATP通道却仍然未知。
2022年5月13日,北京大学未来技术学院分子医学研究所、生命科学联合中心、国家生物医学成像中心陈雷研究组在Nature Communications杂志发表了题为“Structural identification of vasodilator binding sites on the SUR2 subunit”的论文,该研究解析了SUR2与两种血管扩张剂(P1075和Lev)复合物的高分辨结构,揭示了这类小分子药物与SUR2的相互作用模式。
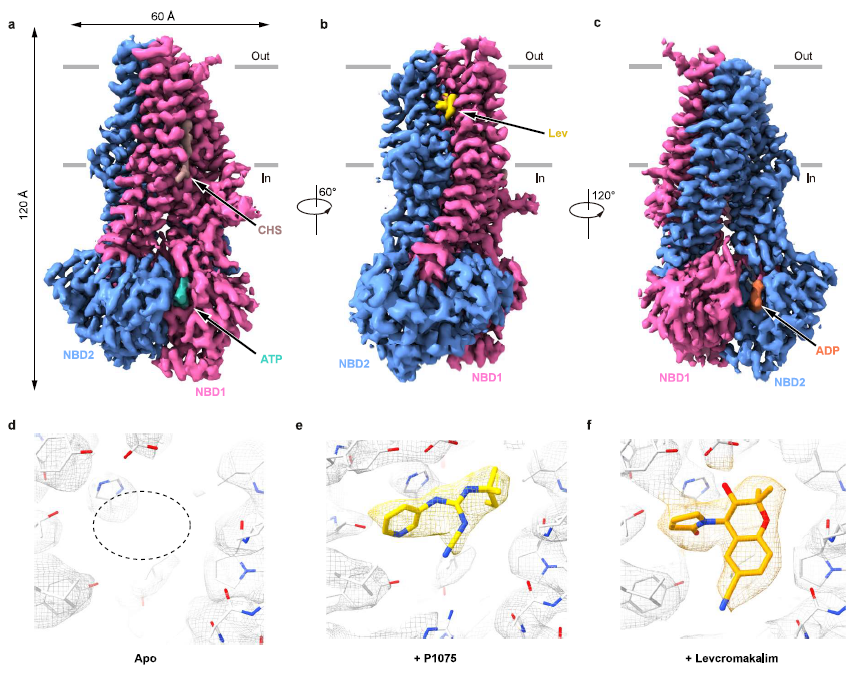
图1:KCO与SUR2复合体结构
根据复合体结构,陈雷研究组发现KCO的结合位点(即命名为KCOS),位于SUR2亚基的跨膜螺旋之间(TM10、TM11、TM12、TM14和TM17)。P1075和Lev结合在SUR2相同位置上,这与P1075、尼可地尔和二氮嗪在SUR2B上竞争性结合行为一致8。
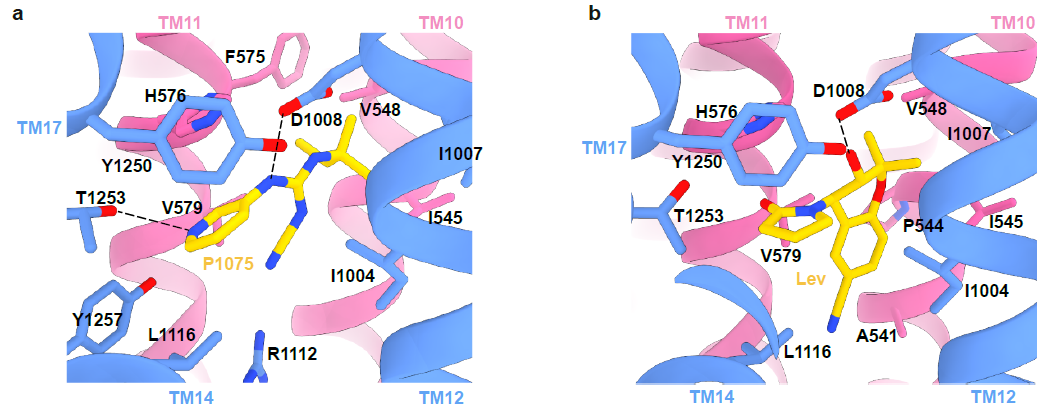
图2:KATP通道开放剂P1075和Lev结合位点
此外,药物复合体结构揭示了SUR2选择性KCO的结构和活性的关系。P1075 是吡那地尔的类似物,对SUR2亚基具有高亲和力。P1075的二甲基丙基替换了吡那地尔的三甲基丙基,使得P1075与由I545、V548、V579和I1004形成的疏水口袋相互作用更强进而显著提高药物激活效果9。作者通过突变实验进一步证明,SUR2的I1004和T1253对于P1075和Lev的激活效果都很重要,将氨基酸突变为SUR1上对应的残基(I1004L 和 T1253M)导致药物激活效果减弱。在Mg-ADP与SUR2的NBD2结合后NBD的二聚化,进而引起TMD1和TMD2的闭合。KATP开放剂与TMD1和TMD2相互作用,促进TMD进入闭合状态,表明Mg核苷酸和KATP开放剂在激活KATP通道时存在正协同作用10,11。
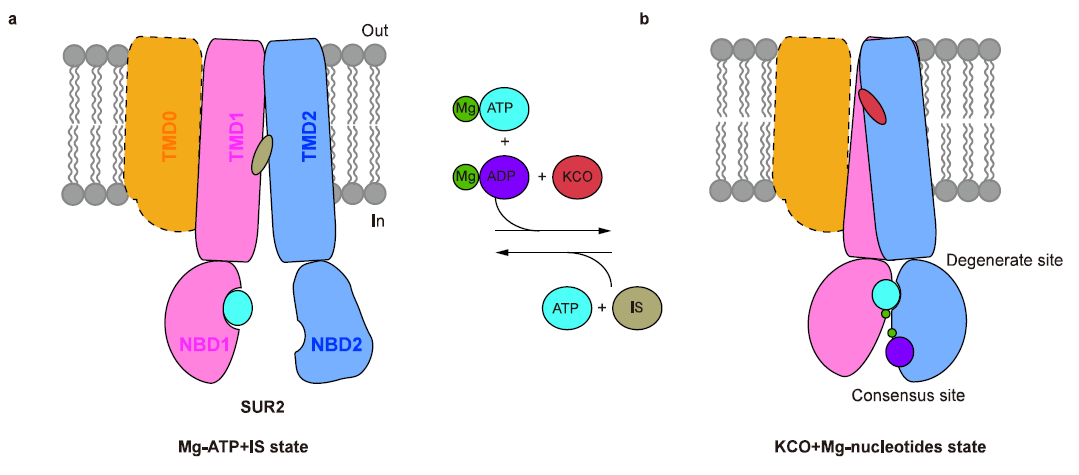
图3:KCO激活SUR2 的模型
此前对胰岛KATP(SUR1-Kir6.2)的结构研究表明:SUR1的NBD二聚化构象与KATP通道激活相关12-15,而SUR1的NBD分离的朝内构象与KATP抑制相关13,16-18。本研究工作发现含有SUR2的KATP通道具有类似的现象,表明SUR1型和SUR2型KATP通道有共同激活机制。该研究为设计和优化KATP开放剂用于治疗相关疾病打下了基础。
本项研究的第一作者为北京大学前沿交叉学科研究院CLS项目博士研究生丁典,陈雷研究员为通讯作者,博士后吴惊香参与了数据收集。晶泰科技的段新丽,马松龄和赖力鹏完成了包括分子动力学模拟在内的计算过程。本工作获得国家自然科学基金委、生命科学联合中心以及北京大学李革-赵宁生命科学青年研究基金的经费支持。该工作冷冻电镜样品制备、筛选和采集在北京大学冷冻电镜平台和北京大学电镜室完成,得到了李雪梅、郭振玺、秦昌东、邵博、裴霞和王国鹏等人的帮助。该项目的数据处理获得了北京大学CLS计算平台及未名超算平台的硬件和技术支持。
论文链接:
https://www.nature.com/articles/s41467-022-30428-y
参考文献:
1. Nichols, C. G. KATP channels as molecular sensors of cellular metabolism. Nature 440, 470-476, doi:10.1038/nature04711 (2006).
2. Pipatpolkai, T., Usher, S., Stansfeld, P. J. & Ashcroft, F. M. New insights into KATP channel gene mutations and neonatal diabetes mellitus. Nat Rev Endocrinol 16, 378-393, doi:10.1038/s41574-020-0351-y (2020).
3. Bienengraeber, M. et al. ABCC9 mutations identified in human dilated cardiomyopathy disrupt catalytic KATP channel gating. Nat. Genet. 36, 382-387, doi:10.1038/ng1329 (2004).
4. Harakalova, M. et al. Dominant missense mutations in ABCC9 cause Cantu syndrome. Nat. Genet. 44, 793-796, doi:10.1038/ng.2324 (2012).
5. van Bon, B. W. et al. Cantu syndrome is caused by mutations in ABCC9. Am. J. Hum. Genet. 90, 1094-1101, doi:10.1016/j.ajhg.2012.04.014 (2012).
6. Jahangir, A. & Terzic, A. K(ATP) channel therapeutics at the bedside. J. Mol. Cell. Cardiol. 39, 99-112, doi:10.1016/j.yjmcc.2005.04.006 (2005).
7. Roy Chowdhury, U., Dosa, P. I. & Fautsch, M. P. ATP sensitive potassium channel openers: A new class of ocular hypotensive agents. Exp. Eye Res. 158, 85-93, doi:10.1016/j.exer.2016.04.020 (2017).
8. Bray, K. M. & Quast, U. A specific binding site for K+ channel openers in rat aorta. J. Biol. Chem. 267, 11689-11692 (1992).
9. Hambrock, A. et al. ATP-Sensitive K+ channel modulator binding to sulfonylurea receptors SUR2A and SUR2B: opposite effects of MgADP. Mol. Pharmacol. 55, 832-840 (1999).
10. Schwanstecher, M. et al. Potassium channel openers require ATP to bind to and act through sulfonylurea receptors. EMBO J. 17, 5529-5535, doi:10.1093/emboj/17.19.5529 (1998).
11. Reimann, F., Gribble, F. M. & Ashcroft, F. M. Differential response of K(ATP) channels containing SUR2A or SUR2B subunits to nucleotides and pinacidil. Mol. Pharmacol. 58, 1318-1325, doi:10.1124/mol.58.6.1318 (2000).
12. Li, N. et al. Structure of a Pancreatic ATP-Sensitive Potassium Channel. Cell 168, 101-110 e110, doi:10.1016/j.cell.2016.12.028 (2017).
13. Wu, J. X. et al. Ligand binding and conformational changes of SUR1 subunit in pancreatic ATP-sensitive potassium channels. Protein Cell 9, 553-567, doi:10.1007/s13238-018-0530-y (2018).
14. Wang, M. et al. Structural insights into the mechanism of nucleotide regulation of pancreatic KATP channel. 2021.2011.2029.470334, doi:10.1101/2021.11.29.470334 %J bioRxiv (2021).
15. Zhao, C. & MacKinnon, R. Molecular structure of an open human KATP channel. Proc. Natl. Acad. Sci. U. S. A. 118, doi:10.1073/pnas.2112267118 (2021).
16. Martin, G. M., Kandasamy, B., DiMaio, F., Yoshioka, C. & Shyng, S. L. Anti-diabetic drug binding site in a mammalian KATP channel revealed by Cryo-EM. Elife 6, doi:10.7554/eLife.31054 (2017).
17. Martin, G. M. et al. Cryo-EM structure of the ATP-sensitive potassium channel illuminates mechanisms of assembly and gating. Elife 6, doi:10.7554/eLife.24149 (2017).
18. Ding, D., Wang, M., Wu, J. X., Kang, Y. & Chen, L. The Structural Basis for the Binding of Repaglinide to the Pancreatic KATP Channel. Cell Rep 27, 1848-1857 e1844, doi:10.1016/j.celrep.2019.04.050 (2019).