

Protein & Cell | 李丹/彭小忠/杨俊涛/苏昭铭发现SARS-CoV-2干扰应激颗粒稳态并促进蛋白病理性相变聚集
日期:2022-04-09 浏览次数:4526
来源:BioArt
应激条件下蛋白质通过相互作用发生液-液相分离(Liquid-liquid phase separation,LLPS)驱动应激颗粒(Stress granule,SG)的形成。SG中的关键致病蛋白TDP-43,FUS及hnRNPA1等的相分离调控紊乱与神经退行性疾病的发病机制密切相关【1】。研究发现,多种病毒,如H1N1流感病毒、疱疹病毒等,在侵染宿主时能入侵中枢神经系统,引起蛋白稳态失调及炎症反应,并与阿尔兹海默病、帕金森病等神经退行性疾病的发病相关【2,3】。近期,对于SARS-CoV-2临床研究揭示部分新冠感染患者的大脑存在脑萎缩现象【4】。另外,SARS-CoV-2侵染会引起脑部炎症反应【5,6】,与神经退行性疾病和精神类疾病的发病存在相关性【7】。
2022年4月6日,上海交通大学李丹课题组、中国医学科学院医学生物学研究所彭小忠课题组、北京协和医学院杨俊涛课题组以及四川大学苏昭明课题组合作在Protein & Cell在线发表题为 SARS-CoV-2 impairs the disassembly of stress granules and promotes ALS-associated amyloid aggregation 的研究成果,发现SARS-CoV-2的核衣壳(Nucleocapsid, N)蛋白在病毒入侵宿主细胞后能进入SG并抑制其动态解聚过程。更为重要的是,N蛋白能促进SG中的RNA结合蛋白TDP-43,FUS及hnRNPA1等的病理性液-固相转变,导致其发生淀粉样聚集,扰乱细胞的蛋白相分离稳态。该工作为理解外源病毒干扰和破坏宿主蛋白相分离提供了启示。
SARS-CoV-2病毒由多种功能性蛋白和RNA组成,其中N蛋白在病毒复制和组装过程中发挥重要作用。据报道,SARS-CoV-2的N蛋白可以与多种SG蛋白发生相互作用【8, 9】,暗示其在调控SG中的潜在作用。在本工作中,研究人员利用SARS-CoV-2病毒侵染HeLa和Vero等哺乳动物细胞,发现SARS-CoV-2 N蛋白在受感染的细胞中表达,并在急性应激条件下被SG招募(图1,A)。同时,N蛋白在进入到宿主细胞SG后显著抑制了应激恢复过程中SG的动态解聚。进一步研究发现,外源转染的N蛋白进入SG后显著减弱了SG的液态流动性,从而抑制受感染细胞中SG的动态解聚过程。值得注意的是,N蛋白自身具有很强的液-液相分离能力,这可能是N 蛋白参与SG组装的驱动因素。另外,长程刺激条件下,受病毒侵染的细胞SG的自发清除效率明显提高,其可能由病毒引发的细胞自噬介导。
接下来,研究人员对N蛋白影响SG解聚的机理进行了研究。之前的蛋白质组学研究发现N蛋白与FUS及hnRNPA1等SG相关蛋白存在相互作用【9】。这类RNA结合蛋白(RNA-binding protein, RBP)能够通过液-液相分离参与SG的组装,而相分离调控紊乱会导致蛋白的液-固相转变及SG的固化,该过程与渐冻人症(Amyotrophic lateral sclerosis,ALS)等疾病的发病密切相关[1]。本研究发现N蛋白能够与FUS、hnRNPA1、TDP-43等ALS相关蛋白发生共分相形成液滴,并进一步加速这蛋白的液-固相转变。其中,N蛋白能显著加速FUS病理性纤维的产生(图1,B);N蛋白通过SARS-CoV-2的侵染能够显著加速FUS-P525L(ALS致病性突变)在细胞中的病理聚集(图1,C)。核磁共振研究显示N蛋白能够与介导FUS及TDP-43相分离及异常聚集的LCD(low-complexity domain)发生直接相互作用,并进一步促进LCD产生纤维状聚集。
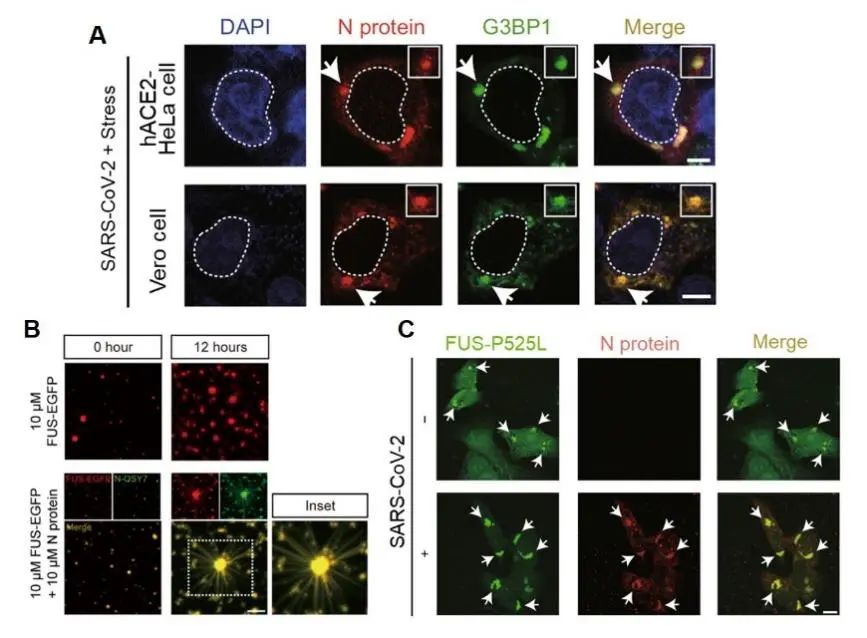
图1. N蛋白与SG共定位并加速SG组分FUS蛋白产生异常聚集
综上,本研究发现SARS-CoV-2能够通过其N蛋白干扰宿主细胞中的SG动态解聚,并且诱导加速TDP-43和FUS等ALS相关蛋白的液-固相转化及病理聚集。本工作为理解SARS-CoV-2入侵对无膜细胞器稳态及蛋白质相分离的影响提供了基础(图2);为研究SARS-CoV-2与神经退行性疾病的潜在关系提供了新的视角。需要特别指出的是,SARS-CoV-2感染能否促进ALS等神经退行性疾病的病理进程还需后续大量的流行病学调查和临床实验来研究和论证。

图2. SARS-CoV-2与宿主SG相互作用示意图。
原文链接:https://doi.org/10.1007/s13238-022-00905-7
1. Wolozin, B., and Ivanov, P. (2019). Stress granules and neurodegeneration. Nat Rev Neurosci 20, 649-666.
2. Readhead, B., Haure-Mirande, J.V., Funk, C.C., Richards, M.A., Shannon, P., Haroutunian, V., Sano, M., Liang, W.S., Beckmann, N.D., Price, N.D., et al. (2018). Multiscale Analysis of Independent Alzheimer's Cohorts Finds Disruption of Molecular, Genetic, and Clinical Networks by Human Herpesvirus. Neuron 99, 64-82 e67.
3. Marreiros, R., Muller-Schiffmann, A., Trossbach, S.V., Prikulis, I., Hansch, S., Weidtkamp-Peters, S., Moreira, A.R., Sahu, S., Soloviev, I., Selvarajah, S., et al. (2020). Disruption of cellular proteostasis by H1N1 influenza A virus causes alpha-synuclein aggregation. Proc Natl Acad Sci U S A 117, 6741-6751.
4. Douaud, G., Lee, S., Alfaro-Almagro, F., Arthofer, C., Wang, C., McCarthy, P., Lange, F., Andersson, J.L.R., Griffanti, L., Duff, E., et al. (2022). SARS-CoV-2 is associated with changes in brain structure in UK Biobank. Nature.
5. Gialluisi, A., de Gaetano, G., and Iacoviello, L. (2020). New challenges from Covid-19 pandemic: an unexpected opportunity to enlighten the link between viral infections and brain disorders? Neurological Sciences 41, 1349-1350.
6. Bhaskar, S., Sinha, A., Banach, M., Mittoo, S., Weissert, R., Kass, J.S., Rajagopal, S., Pai, A.R., and Kutty, S. (2020). Cytokine Storm in COVID-19—Immunopathological Mechanisms, Clinical Considerations, and Therapeutic Approaches: The REPROGRAM Consortium Position Paper. Frontiers in Immunology 11.
7. Bauer, L., Laksono, B.M., de Vrij, F.M.S., Kushner, S.A., Harschnitz, O., and van Riel, D. (2022). The neuroinvasiveness, neurotropism, and neurovirulence of SARS-CoV-2. Trends Neurosci.
8. Moosa, M.M., and Banerjee, P.R. (2020). Subversion of host stress granules by coronaviruses: Potential roles of pi-rich disordered domains of viral nucleocapsids. J Med Virol.
9. Gordon, D.E., Jang, G.M., Bouhaddou, M., Xu, J., Obernier, K., White, K.M., O'Meara, M.J., Rezelj, V.V., Guo, J.Z., Swaney, D.L., et al. (2020). A SARS-CoV-2 protein interaction map reveals targets for drug repurposing. Nature 583, 459-468.