

孙飞/张传茂合作揭示核孔复合体外环精细结构
日期:2022-01-13 浏览次数:10337
来源:BioArt
生物进化的一大标志性事件是细胞内出现不同膜结构包围的细胞器,其中最重要的当属细胞核。细胞核将遗传物质的储存,转录与蛋白质的表达等生命过程分隔开,保证了细胞内遗传物质的稳定。细胞核膜上存在唯一的沟通核质、胞质的物质运输的孔道,称为核孔复合体。核孔复合体由约30多种核孔蛋白组成,相对于核孔中央孔道按八重对称性排列为空心圆柱状,直径约120纳米,高60~80纳米,分子质量可达66~120兆道尔顿。不同的核孔蛋白(Nup)组成一系列亚复合物,这些亚复合物相互之间巧妙连接,分别构成了核孔复合体中的胞质环、内环、腔内环、核质环、胞质纤维、核篮和中央孔道内的透过屏障等部分,再搭建成完整的核孔复合体。除了沟通细胞核质与胞质的物质运输以外,核孔复合体的某些组分还参与了一些复杂的生命活动,如DNA修复,基因表达调控,细胞自噬等等。由于其巨大的尺寸、复杂的生理功能和高度动态性的结构特征,很长时间以来,核孔复合体的精细结构研究始终是结构生物学领域的一大难点。在过去相当长的一段时期,核孔复合体大部分结构生物学研究都是采用冷冻电镜电子断层成像技术,最终分辨率在20埃左右,研究对象集中于相对刚性的胞质环、内环、核质环和腔内环等部分。
最近几个月,在Nature,Science,Cell以及预印本平台bioRxiv上,共有来自于国内外7个不同团队的11篇关于核孔复合体的结构生物学研究陆续发表,掀起了一股核孔复合体研究的浪潮,标志着核孔复合体精细结构研究取得了重大进展。这些工作以酵母源,人源和爪蟾源核孔复合体作为研究对象,侧重于核孔复合体的不同方面进行了深入细致的研究。10月和11月的两篇Nature与Science杂志文章侧重于研究核孔复合体与细胞生理状态之间的联系,分别通过对人源和酵母源核孔复合体在原位的结构研究,发现核孔复合体在生理状态下存在不同的开合状态。发表在bioRxiv上的几篇文章则侧重于核孔复合体的精细结构。其中,Martin Beck和Jan Kosinski研究组联合,以纳米级分辨率的人源核孔复合体高分辨结构为基础,利用AlphaFold和同源建模软件发表了近乎于完整的人源核孔复合体模型。施一公研究组和隋森芳研究组分别对爪蟾和酵母核孔复合体的内环高分辨率结构进行了研究。2022年1月Cell杂志文章利用综合技术手段研究了酵母核孔复合体在分离纯化状态下的高分辨率结构和在原位生理状态下的相对低分辨率结构,阐明了酵母核孔复合体的模块化组装形式及其与生理功能状态的关系。这些研究成功极大的加深了人们对核孔复合体组装机制的理解。
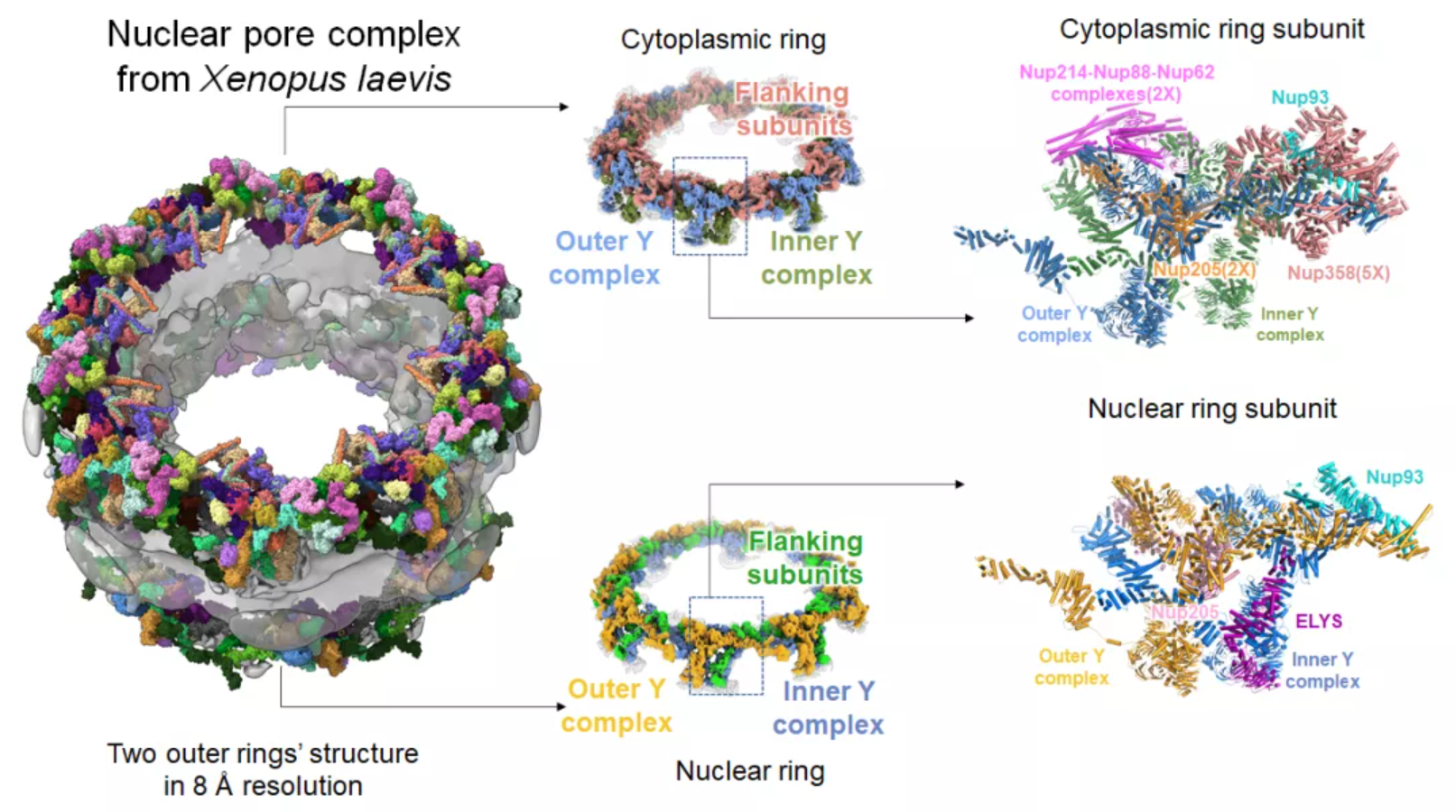
2022年1月11日,中国科学院生物物理研究所孙飞研究组与北京大学生命科学学院张传茂研究组联合在Protein & Cell杂志发表了题为“8 Å structure of the outer rings of the Xenopus laevis nuclear pore complex obtained by cryo-EM and AI”的研究论文,获得爪蟾核孔复合体外环(即胞质环和核质环)的亚纳米级分辨率的冷冻电镜三维结构和原子模型。由于电子断层成像技术天然受限于图像相对的低信噪比所带来的一系列问题,在该研究中,研究者采用冷冻电镜单颗粒技术对手动铺展在电镜载网上的爪蟾卵母细胞核膜进行成像。为了克服铺展开的核膜引入的取向优势的问题,研究者采用了倾转样品台成像和改变铺展核膜的方式以取得核孔复合体侧向投影的特殊方法,最终得到了近乎于各向同性分辨率的爪蟾核孔复合体外环的重构结果。
前人的研究虽然说明了爪蟾核孔复合体外环基本骨架的成分及其相对位置,但仍有一些缺陷,比如结构模型还不完整(有的亚基整个缺失,有的亚基还是截短体),很多亚基之间间隔较远,没有能够紧密结合等等。有鉴于此,该研究利用AlphaFold2和爪蟾核孔蛋白序列预测了所有核孔蛋白的结构,利用手动建模方式结合半自动化的原子模型修正软件对已经鉴定出的核孔外环组分进行逐个建模。他们首先搭建得到完整外环骨架结构,即Y字形复合体的结构模型;在此基础上,将已经模建部分的密度图减去,其余的部分继续利用AlphaFold2预测得到的高度可信的核孔蛋白结构配合电镜三维重构体中的二级结构信息,将其余部分的亚基进行依次模建。
利用上述方法,研究者最终搭建得到爪蟾核孔复合体外环的目前最完整的结构模型,清楚解释了Y字形复合体不同组分间的相互作用模式(图1)。进一步地,研究者准确指认了在爪蟾核孔复合体胞质环和核质环的每个不对称单元中,存在2个或是1个Nup205,而不是之前认为的Nup188,作为不同Y字复合体之间的粘合剂。同样的,在每个胞质环或核质环不对称单元上,都存在一个Nup93作为桥接的组分稳定不同Y字复合体的根部区域。在每个胞质环不对称单元上,有两个Nup214复合体组成mRNP出核平台,5个Nup358亚基紧紧环绕胞质环Y字复合体的根部区域,一方面稳定了胞质环不对称单元,另一方面组成胞质纤维。在核质侧,每个不对称单元有且仅有一个ELYS,粘附在内Nup160背后,在有丝分裂后期起始核孔复合体组装。该研究关注于爪蟾核孔复合体亚纳米分辨率的外环结构,揭示了其上众多亚基的空间位置和相互作用关系,使得核孔复合体外周环的结构模型得到了完整的解析,为全面揭示核孔复合体的结构与功能奠定了基础。
原文链接:
https://link.springer.com/content/pdf/10.1007/s13238-021-00895-y.pdf
参考文献:
1. D. H. Lin et al., Architecture of the symmetric core of the nuclear pore. Science 352, aaf1015 (2016).
2. T. Stuwe et al., Nuclear pores. Architecture of the nuclear pore complex coat. Science 347, 1148-1152 (2015).
3. T. Stuwe et al., Architecture of the fungal nuclear pore inner ring complex. Science 350, 56-64 (2015).
4. C. E. Zimmerli et al., Nuclear pores dilate and constrict in cellulo. Science 374, eabd9776 (2021).
5. M. Allegretti et al., In-cell architecture of the nuclear pore and snapshots of its turnover. Nature 586, 796-800 (2020).
6. J. Kosinski et al., Molecular architecture of the inner ring scaffold of the human nuclear pore complex. Science 352, 363-365 (2016).
7. A. von Appen et al., In situ structural analysis of the human nuclear pore complex. Nature 526, 140-143 (2015).
8. S. J. Kim et al., Integrative structure and functional anatomy of a nuclear pore complex. Nature 555, 475-482 (2018).
9. J. Fernandez-Martinez et al., Structure and Function of the Nuclear Pore Complex Cytoplasmic mRNA Export Platform. Cell 167, 1215-1228.e1225 (2016).
10. F. Alber et al., The molecular architecture of the nuclear pore complex. Nature 450, 695-701 (2007).
11. F. Alber et al., Determining the architectures of macromolecular assemblies. Nature 450, 683-694 (2007).
12. G. Huang et al., Structure of the cytoplasmic ring of the Xenopus laevis nuclear pore complex by cryo-electron microscopy single particle analysis. Cell Res 30, 520-531 (2020).
13. Y. Zhang et al., Molecular architecture of the luminal ring of the Xenopus laevis nuclear pore complex. Cell Res 30, 532-540 (2020).
14. C. W. Akey et al., Comprehensive structure and functional adaptations of the yeast nuclear pore complex. Cell,(2022).
15. L. Tai et al., 8 Å structure of the outer rings of the Xenopus laevis nuclear pore complex obtained by cryo-EM and AI. Protein & Cell, (2022).
16. G. Huang et al., Cryo-EM structure of the Inner Ring from Xenopus laevis Nuclear Pore Complex. bioRxiv, 2021.11.13.468242 (2021).
17. Z. Li et al., Near atomic structure of the inner ring of the Saccharomyces cerevisiae nuclear pore complex. bioRxiv, 2021.11.26.470097 (2021).
18. S. Mosalaganti et al., Artificial intelligence reveals nuclear pore complexity. bioRxiv, 2021.10.26.465776 (2021).
19. S. Petrovic et al., Architecture of the linker-scaffold in the nuclear pore. bioRxiv, 2021.10.26.465796 (2021).
20. C. J. Bley et al., Architecture of the cytoplasmic face of the nuclear pore. bioRxiv, 2021.10.26.465790 (2021).
21. H. Ren et al., Determining the architecture of nuclear ring of Xenopus laevis nuclear pore complex using integrated approaches. bioRxiv, 2021.11.10.468004 (2021).