

Mol Biol Evol|吴爱平课题组揭示流感病毒抗原变异的关键分子机制
日期:2019-03-13 浏览次数:8522
季节性流感病毒的反复流行和变异,给整个人类社会带来了巨大的经济损失和疾病负担。根据世界卫生组织(WHO)的估算,每年流感病毒感染的全球人群高达1亿,其中300-500万人显著患病,并且有29-65万的患者死亡。目前,最有效的流感防控策略依然是对人群进行疫苗接种。然而,由于流感病毒主要表面抗原蛋白HA上频繁的位点突变,导致突变病毒更容易逃避宿主免疫,从而发生显著的抗原变异,使得疫苗保护失效。因此,如果能识别出病毒抗原变异的决定性位点突变,掌握这些关键位点突变的结构、功能和进化特征等,将为流感病毒的变异监测、趋势预警和疫苗株选择提供非常重要的指导。

图1:抗原决定性位点识别方法RECDS的方法学流程
针对这一重要科学问题,吴爱平课题组开发了一个基于机器学习的抗原决定性位点识别方法RECDS(图1),通过整合病毒序列、结构和进化信息,系统地度量了流感病毒HA蛋白上每个位点在整个流感病毒进化历史中的抗原贡献,识别出了甲型流感H3N2和H1N1病毒抗原变异过程中最关键的10-15个决定性突变位点(图2)。他们发现,流感病毒H3N2的抗原类变异决定性位点主要分布在HA蛋白的受体结合区(RBD)附近,而H1N1除了分布在RBD区外,还分布在另外一个残留酯酶区域(ED),这表明两种亚型病毒既采用了一致的突变策略,又有自身特异性的变异机制(图2)。这也一定程度上解释了为什么甲型H3N2病毒抗原变异快而H1N1病毒抗原变异相对较慢。

图2:流感病毒H3N2和H1N1的抗原决定性位点的结构分布特征
通过结合病毒进化信息,该研究还进一步刻画出了每个关键位点在抗原变异过程中的突变类型和历史(图3),为病毒位点突变和抗原变异的对应监测提供了直接支持。此外,从病毒角度,他们系统描绘出了流感病毒抗原变异过程中的整体关键位点突变轨迹和支配规律(图4),为疫苗株推荐提供了关键位点靶标。
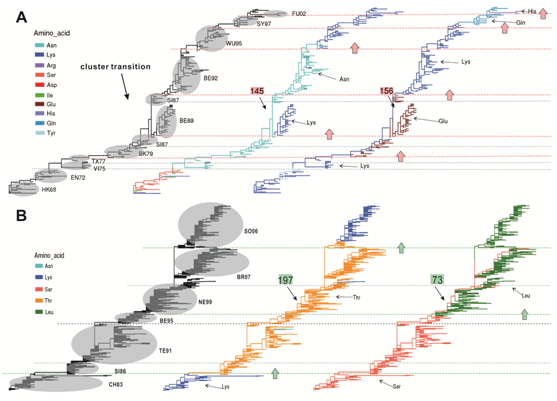
图3:代表性抗原决定性位点的进化分析
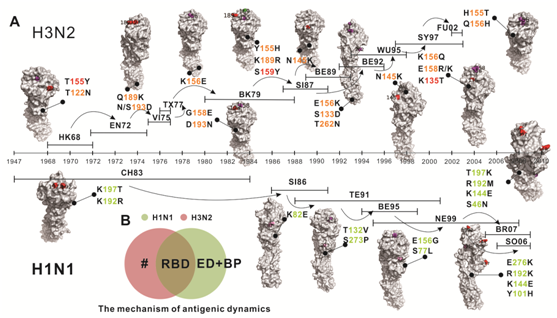
图4:季节性流感H3N2和H1N1抗原决定性位点的突变路径
上述成果已于2019年3月9日发表在国际著名期刊Molecular Biology and Evolution上,题为“Cluster-transition determining sites underlying the antigenic evolution of seasonal influenza viruses”。中国医学科学院苏州系统医学研究所吴爱平课题组的权丽君博士为该项工作的第一作者,吴爱平研究员为通讯作者。该研究得到了国家重点研发计划、国防科技创新特区项目、医科院医学与健康科技创新工程、国家和江苏省自然科学基金以及江苏省六大人才高峰等项目的共同支持。
文章链接: https://academic.oup.com/mbe/advance-article-abstract/doi/10.1093/molbev/msz050/5372682